生信 R语言
2024-01-07 17:21:47
11.芯片表达矩阵下游分析
?rm(list = ls())#清除所有变量
options(stringsAsFactors = F)
#BiocManager::install("CLL")
suppressPackageStartupMessages(library(CLL))
data("sCLLex")
sCLLex## ExpressionSet (storageMode: lockedEnvironment)
## assayData: 12625 features, 22 samples
## element names: exprs
## protocolData: none
## phenoData
## sampleNames: CLL11.CEL CLL12.CEL ... CLL9.CEL (22 total)
## varLabels: SampleID Disease
## varMetadata: labelDescription
## featureData: none
## experimentData: use 'experimentData(object)'
## Annotation: hgu95av2exprSet=exprs(sCLLex)
samples=sampleNames(sCLLex)
pData=pData(sCLLex)
#没有代码,暂未做12.RNA-seq 表达矩阵差异分析
rm(list = ls())
options(stringsAsFactors = F)
library(airway)
data("airway") #加载数据
exprSet=assay(airway)
colnames(exprSet)
group_list=colData(airway)[,3]
exprSet=exprSet[apply(exprSet, 1,function(x)sum(x>1)>5),]
table(group_list)
group_list
exprSet[1:4,1:4]
boxplot(exprSet)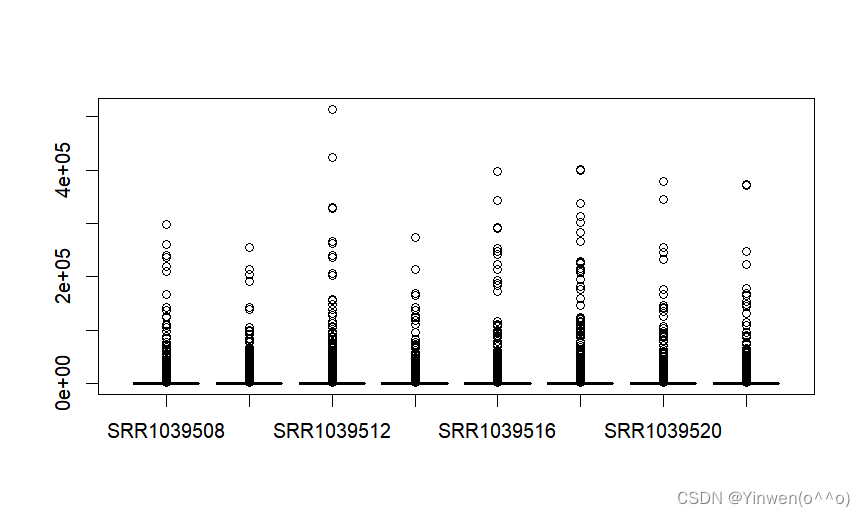
boxplot(log(exprSet+1))
dev.off()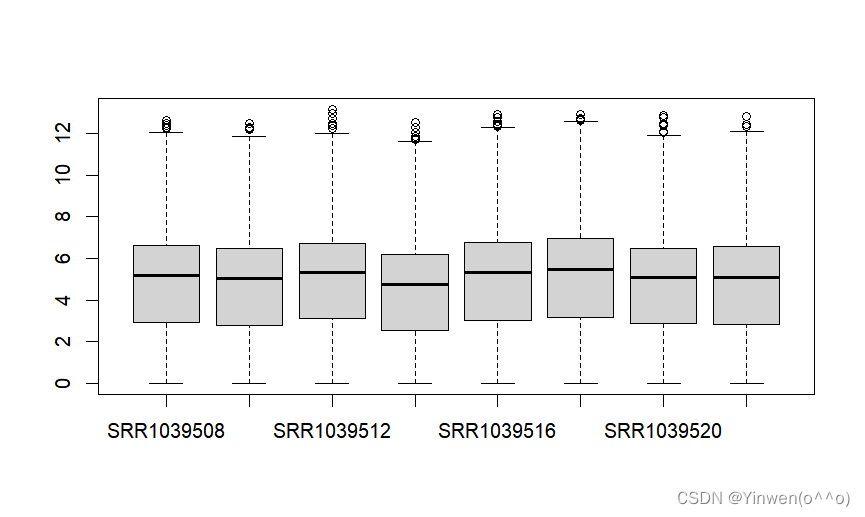
文章来源:https://blog.csdn.net/m0_56780122/article/details/135430981
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:veading@qq.com进行投诉反馈,一经查实,立即删除!
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:veading@qq.com进行投诉反馈,一经查实,立即删除!