代码+视频,手把手教你R语言使用scitb包绘制倾向性评分匹配后的标准化平均差图
什么是倾向性评分匹配?倾向评分匹配(Propensity Score Matching,简称PSM)是一种统计学方法,用于处理观察研究(Observational Study)的数据,在SCI文章中应用非常广泛。在观察研究中,由于种种原因,数据偏差(bias)和混杂变量(confounding variable)较多,倾向评分匹配的方法正是为了减少这些偏差和混杂变量的影响,以便对实验组和对照组进行更合理的比较。
为什么需要做倾向评分匹配?
我们知道RCT的证据力度高,是因为对患者进行了严格的筛选。我们的回顾性研究都是过去的数据,很难像RCT一样进行严格的筛选出两组患者基线相近的基础资料,但我们可以通过倾向评分匹配把回归性的数据进行筛选,把基线资料相近的患者进行匹配,得到近似RCT的效果。
应用场景
? 1.基线资料不平
? 2.开展病例对照研究病阳性例数较少,如罕见病研究
? 3.将众多混杂因素变为一个变量:倾向值
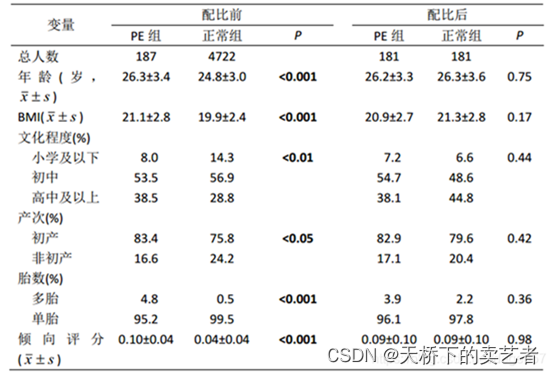
以下为一个实例,没进行匹配前两组患者基线资料相差很大,进行倾向评分匹配后,基线资料近似一致了
标准化平均差SMD是什么?
而在医学研究中,SMD最常用的地方可能是在随机对照试验中,或进行倾向性评分匹配或加权前后,来评估两组组间基线资料的均衡性。

在评估组间的均衡性时,SMD<0.1通常表示均衡性较好,可以认为研究组之间的差异很小。
Scitb包在更新1.7版本后,就可以生成标准化平均差(Standardized mean difference SMD)值了,这样让它多出了一个小技能,绘制倾向性评分匹配后的标准化平均差(Standardized mean difference SMD)可视化图。
下面我来演示一下,先导入数据和R包
library(Matching)
library(scitb)
rhc<-read.csv("E:/r/test/rhc.csv",sep=',',header=TRUE)
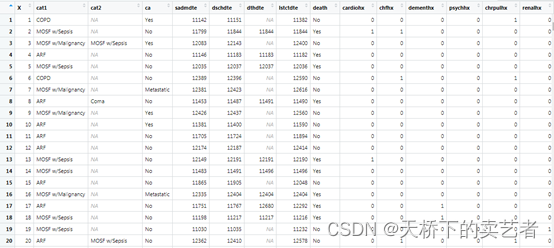
这是一个右心导管介入治疗的数据,主要是介绍了心导管治疗后的生存数据,这里我就不一一介绍了,公众号回复:右心导管数据,可以获得这个数据。我们需要先看一下这个数据的基线情况,先要定义全部变量,分类变量,分层变量
定义全部变量,就是咱们要观察的所有变量
vars <- c("age","sex","race","edu","income","ninsclas","cat1","das2d3pc","dnr1",
"ca","surv2md1","aps1","scoma1","wtkilo1","temp1","meanbp1","resp1",
"hrt1","pafi1","paco21","ph1","wblc1","hema1","sod1","pot1","crea1",
"bili1","alb1","resp","card","neuro","gastr","renal","meta","hema",
"seps","trauma","ortho","cardiohx","chfhx","dementhx","psychhx",
"chrpulhx","renalhx","liverhx","gibledhx","malighx","immunhx",
"transhx","amihx")
定义全部变量中的分类变量
fvars<-c("sex","race","income","ninsclas","cat1","dnr1",
"ca","resp","card","neuro","gastr","renal","meta","hema","seps","trauma","ortho")
定义分层变量
strata<-"swang1"
生成数据的基线表,基线表生成的操作详见上一篇文章《R语言使用scitb包10分钟快速绘制论文基线表》,这里注意一下,参数smd = T一定要有,因为等下咱们要使用smd来绘制
out<-scitb1(vars=vars,fvars=fvars,strata=strata,data=rhc,statistic=T,smd = T)
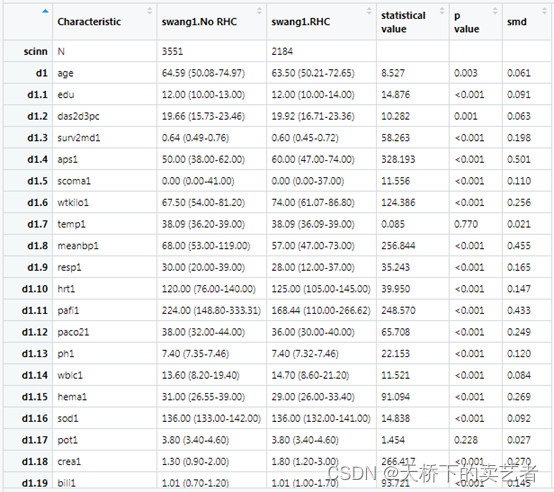
上图咱们可以看到,基线表已经生成了,有很多变量的smd很高,有些P值小于0.05。接下来咱们进行倾向性评分匹配,具体操作可以看文章《R语言基于Matching包进行倾向评分匹配(PSM)》,这里咱们就简单过一下代码了
变量转成因子
rhc$swang1 <- factor(rhc$swang1, levels = c("No RHC", "RHC"))
生成预测模型
psModel <- glm(formula = swang1 ~ age + sex + race + edu + income + ninsclas +
cat1 + das2d3pc + dnr1 + ca + surv2md1 + aps1 + scoma1 +
wtkilo1 + temp1 + meanbp1 + resp1 + hrt1 + pafi1 +
paco21 + ph1 + wblc1 + hema1 + sod1 + pot1 + crea1 +
bili1 + alb1 + resp + card + neuro + gastr + renal +
meta + hema + seps + trauma + ortho + cardiohx + chfhx +
dementhx + psychhx + chrpulhx + renalhx + liverhx + gibledhx +
malighx + immunhx + transhx + amihx,
family = binomial(link = "logit"),
data = rhc)
生成预测概率,有些文章直接就用预测概率来匹配了,我这里用另一种方法来匹配
rhc$pRhc <- predict(psModel, type = "response")
生成没有发生结局的概率
rhc$pNoRhc <- 1 - rhc$pRhc
进行倾向评分匹配
listMatch <- Match(Tr = (rhc$swang1 == "RHC"), # Need to be in 0,1
## logit of PS,i.e., log(PS/(1-PS)) as matching scale
X = log(rhc$pRhc / rhc$pNoRhc),
## 1:1 matching
M = 1,
## caliper = 0.2 * SD(logit(PS))
caliper = 0.2,
replace = FALSE,
ties = TRUE,
version = "fast")
匹配成功后提取匹配的数据
rhcMatched <- rhc[unlist(listMatch[c("index.treated","index.control")]), ]
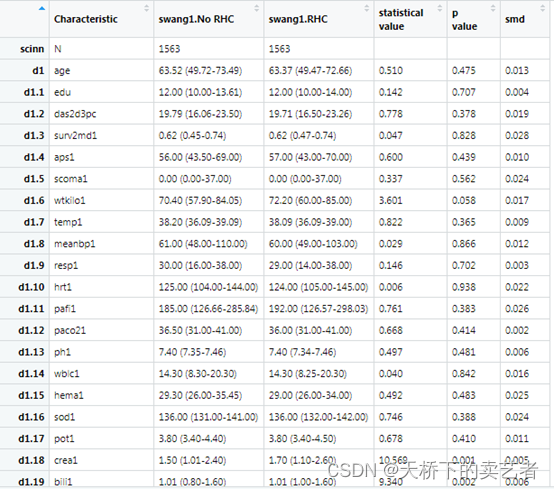
生成匹配后数据的基线表,可以看到匹配后P值变大,smd明显变小
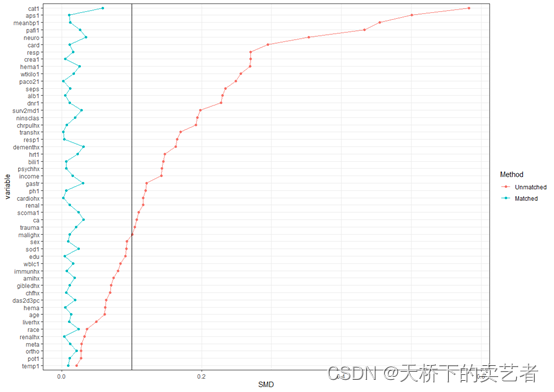
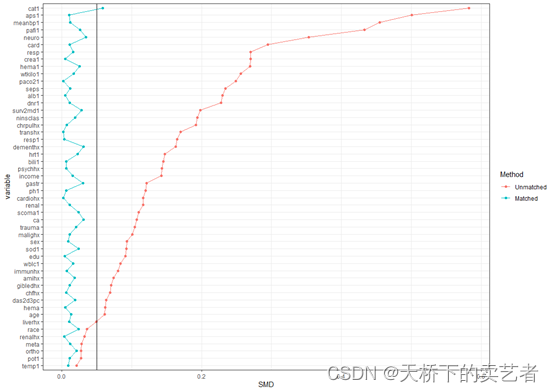
绘制倾向性评分匹配后的标准化平均差(Standardized mean difference SMD)可视化图,vars这里填入你所有的基线资料的变量,Unmatchdata这里填入未匹配前的数据,matchdata这里填入匹配后的数据
plotsmd(vars = vars,unmatchdata = out,matchdata = out1)
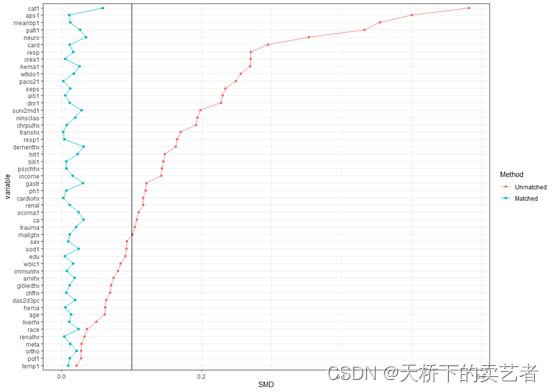
上图可以清晰的看到没匹配后的smd明显小于匹配前的,匹配效果非常好,我设置默认参考线在0.1这个位置,咱们还可以更改一下
plotsmd(vars = vars,unmatchdata = out,matchdata = out1,refline=0.05)
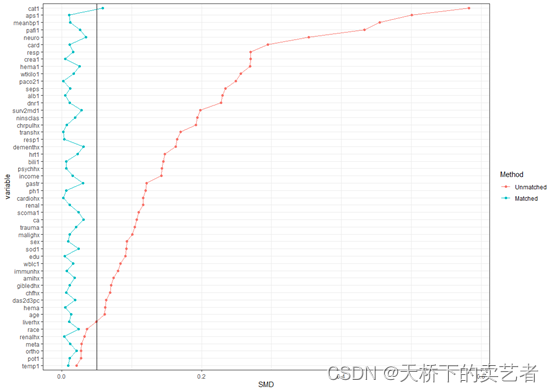
也可以对标题和X轴,Y轴进一步修改
plotsmd(vars = vars,unmatchdata = out,matchdata = out1,refline=0.05,
title="SMD比较图",xlab='SMD值',ylab='变量')
配套视频
代码+视频,手把手教你R语言使用scitb包绘制倾向性评分匹配后的标准化平均差图
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:veading@qq.com进行投诉反馈,一经查实,立即删除!