机器学习 | SVM支持向量机
?
?
?
?
1、核心思想及原理
???????? 针对线性模型中分类两类点的直线如何确定。这是一个ill-posed problem。
???? ? ? 线性模型通过求解各点距离进行投票,可以使用sigmoid函数来求解总损失,转换成一个最优化问题。但是模型泛化能力太差,很容易受到噪声的干扰,直线可能会发生很大变化。
??? ? ?? 所以我们更想让直线靠近中间位置。
????????
????????
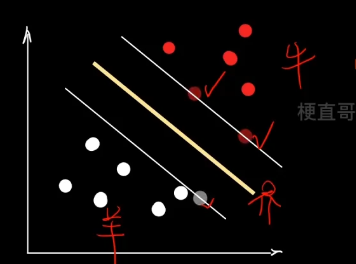
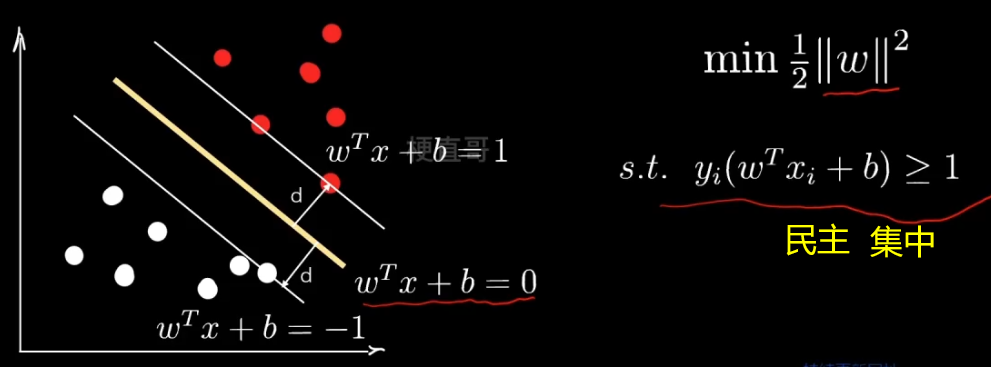
??????? 三个概念:
??????? 支持向量 support vector —— 距离决策边界最近的点
??????? 超平面 hyperplane —— “隔离带”中间的平分线
??????? 间隔 margin —— 最大化margin
????????
??????? 民主集中的思想 ~
?
????????
?
2、线性SVM —— 硬间隔 Hard Margin
?
?(假定数据本身线性可分)
??????? 通过上一小节我们得到优化目标:
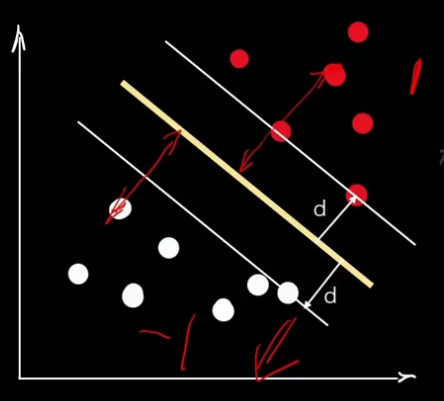
??????? 最大化间隔margin 也就是 最大化距离 d,也就是点到超平面的垂直距离。
??????? 注意此处的距离和线性模型中的距离不同,线性模型中的距离是 yhat-y (斜边)
????????
?
??????? SVM的求解过程和线性方程一样:
??????????????? 先假设一个超平面,
??????????????? 这里的W和X都是高维向量,点到高维空间中的距离有现成公式可以套用:
??????????????? ||w||为w的范数,表示高维空间中长度的概念。
????????????????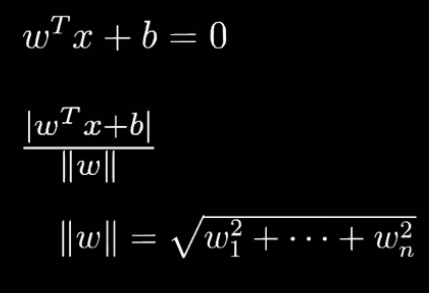
??????????????? 有了这样一个距离公式,隔离带就可以表示为一个分段函数的形式。
????????????????我们用1和-1来表示两个类别:
????????????????
????????????????
??????????????? 现在我们给柿子两边同时除以d 变形,再继续整理做变量代换:
??????????????? 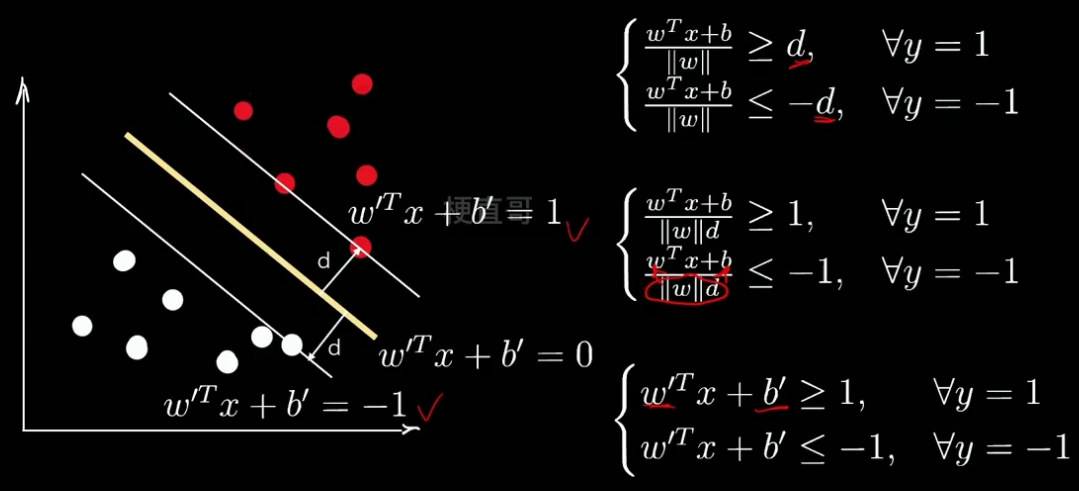
??????????????? 为了方便表示,看着撇多难受,我们再用w和b换回去,
????????????????但此时的w b和一开始推到时的w b,理论上已经不是同一个数了。
????????????????但是存在一个比例关系。在做一个变形:
????????????????
??????????????? 则现在的问题就简化为:
????????????????
???????????????? 这是一个不等式约束的最优化问题。梯度下降只能用于无约束的最优化。
???? ?? ???????? ps:
???????????? ? ? 不等式约束最优化 引入 松弛变量 到等式约束最优化。
???????? ? ????? 等式约束最优化 引入 拉格朗日乘子 到无约束最优化。
?
????????注意,这里的 i 是遍历所有的数据点。但在最小化范数时,我们只用到了支持向量。
????????
???????? 但是如果你相信的人也出了问题呢?
?
3、线性SVM —— 软间隔 Soft Margin
??????? 软间隔要解决的问题:
??????????????? 异常值 outliner??????
??????????????? 支持向量的选择、民主集中思想的弱点
????????????????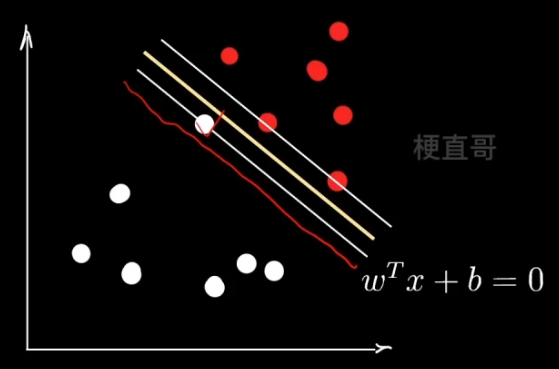
??????? 则需:建立容错机制,提升泛化能力
??????? 那么体现在数学上:允许点越过线一点点 ~
????????
??????? 那么怎么保证 这个减去的值不能太大呢?
????????
??????? 也就是说尽量让所有数据容错值的和最小。让二者取一个平衡。
??????? C 就是一个新的超参数,用来调节两者的权重值。
?
??????? 再看一下这个求和的形式,是不是特别像正则化?其实就可以看成正则化。
????????正则化项是一次的,所以叫L1正则。这里省略了绝对值符号,因为其就是正数。
????????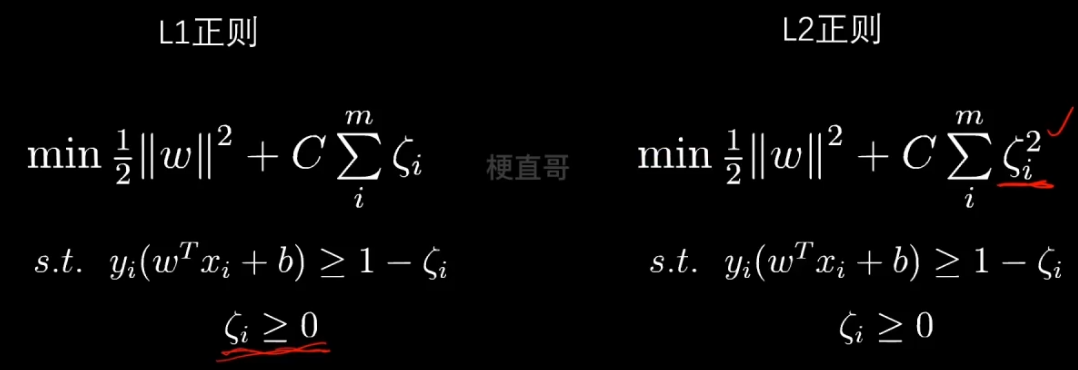
?
?
?4、线性SVM代码实现
?
数据集
import numpy as np
import matplotlib.pyplot as pltfrom sklearn.datasets import make_blobs
x, y = make_blobs(
n_samples=40,
centers=2,
random_state=0
)plt.scatter(x[:,0], x[:,1], c = y)
plt.show()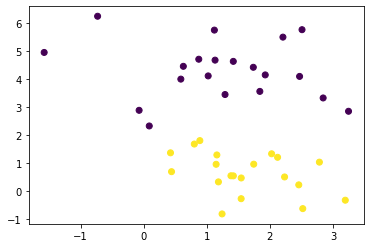
?
sklearn中的线性SVM
from sklearn.svm import LinearSVCclf = LinearSVC(C = 1)clf.fit(x, y)LinearSVC
LinearSVC(C=1)
clf.score(x, y)1.0
def decision_boundary_plot(X, y, clf):
axis_x1_min, axis_x1_max = X[:,0].min() - 1, X[:,0].max() + 1
axis_x2_min, axis_x2_max = X[:,1].min() - 1, X[:,1].max() + 1
x1, x2 = np.meshgrid( np.arange(axis_x1_min,axis_x1_max, 0.01) , np.arange(axis_x2_min,axis_x2_max, 0.01))
z = clf.predict(np.c_[x1.ravel(),x2.ravel()])
z = z.reshape(x1.shape)
from matplotlib.colors import ListedColormap
custom_cmap = ListedColormap(['#F5B9EF','#BBFFBB','#F9F9CB'])
plt.contourf(x1, x2, z, cmap=custom_cmap)
plt.scatter(X[:,0], X[:,1], c=y)
plt.show()decision_boundary_plot(x, y, clf)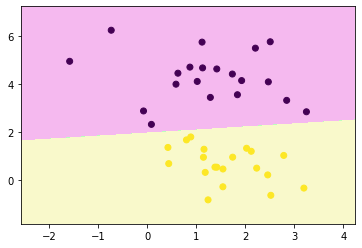
def plot_svm_margin(x, y, clf, ax = None):
from sklearn.inspection import DecisionBoundaryDisplay
DecisionBoundaryDisplay.from_estimator(
clf,
x,
ax = ax,
grid_resolution=50,
plot_method="contour",
colors="k",
levels=[-1, 0, 1],
alpha=0.5,
linestyles=["--", "-", "--"],
)
plt.scatter(x[:,0], x[:,1], c = y)plot_svm_margin(x, y, clf)
plt.show()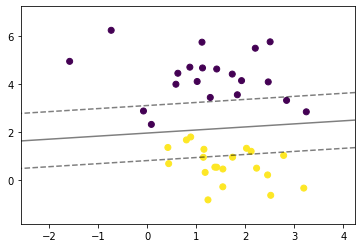
plt.rcParams["figure.figsize"] = (12, 8)
params = [0.1, 1, 10, 100]
for i, c in enumerate(params):
clf = LinearSVC(C = c, random_state=0)
clf.fit(x, y)
ax = plt.subplot(2, 2, i + 1)
plt.title("C: {0}".format(c))
plot_svm_margin(x, y, clf, ax)
plt.show()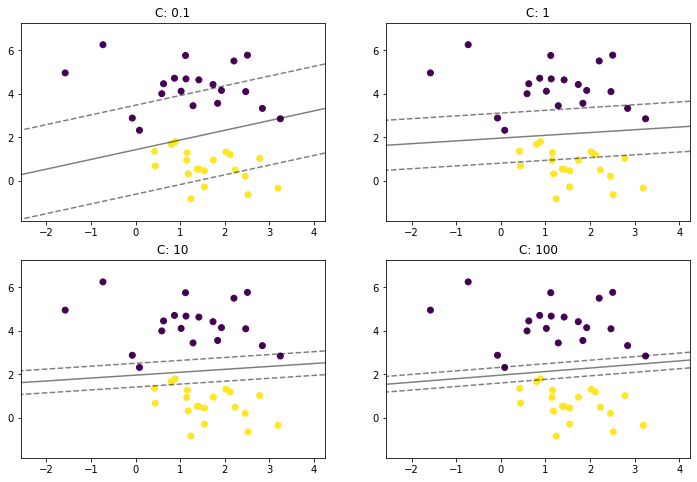
?
多分类
from sklearn import datasets
iris = datasets.load_iris()
x = iris.data
y = iris.target注意SVM中只支持ovr ~
clf = LinearSVC(C = 0.1, multi_class='ovr', random_state=0)
clf.fit(x, y)LinearSVC
LinearSVC(C=0.1, random_state=0)
clf.predict(x)array([0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
2, 1, 1, 1, 2, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 2, 2, 2, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2,
2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 1, 2, 2,
2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2])
?
?
?5、非线性SVM —— 核技巧
?
线性不可分问题
????????
???????? 升维映射
????????
??????? 高维线性可分
????????
?????? 已经有前人在数学上证明:
??????? 如果原始的空间是有限的,也就是说特征数是有限的,
????????那么一定存在一个高位的特征空间能够分割这些样本。
????????
软间隔对偶 dual 问题?
????????
??????? 则现在就要解决右边的问题。
?????????
??????? 套上一个 φ 就可以升维,但是可能会导致维度灾难,所以就要使用核函数。
????????
常用的核函数
????????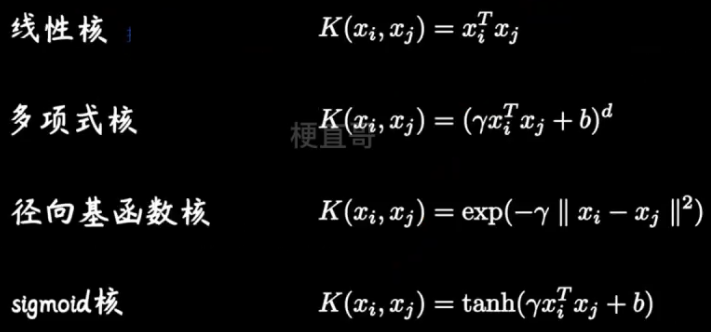
??????? 他们就像一个武器套装,能够方便针对软间隔对偶问题实现升维操作。
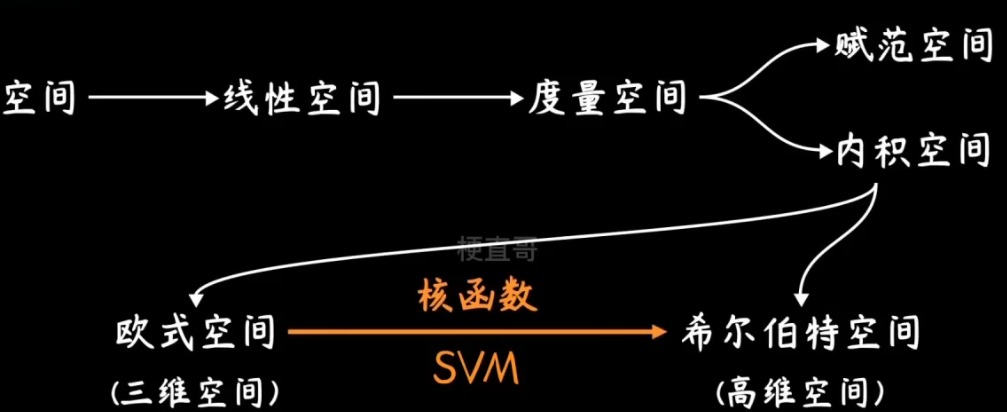
?那么为什么升维就能解决不可分的问题呢?
??????? 希尔伯特空间的元素是函数,因此可以看成无穷维度的向量。
????????
?
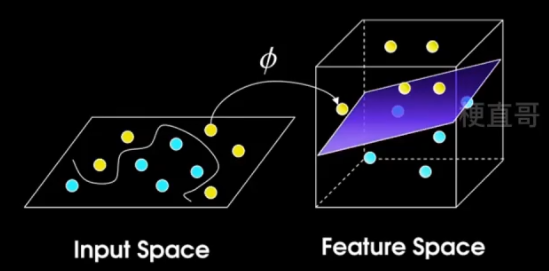
?核技巧的作用
??????? 映射到高维空间
??????? 大大减少了计算量,尤其是对于高维向量的相乘操作,同时也不需要额外存储中间的结果
??????? 不仅限于SVM,实际上 只要存在着 xi ,xj 的最优化问题。
????????
?
?
?6、非线性SVM —— 核函数
?
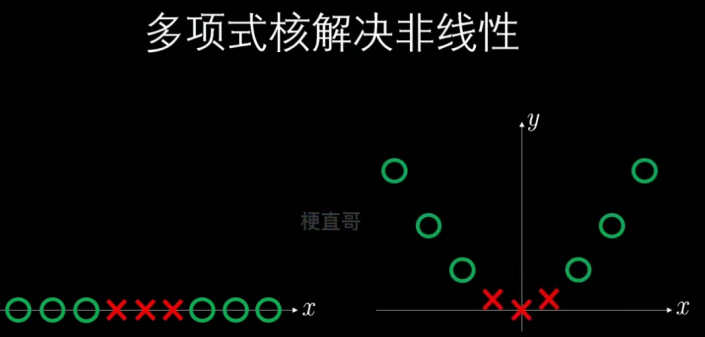
多项式核函数
???????? ?
?
????????以下将说明,为什么这个核函数相当于引入了多项式的特征。
??????? 为了推导方便,此处使用x,y
????????????????
????????????????
????????
高斯核函数
????????
??????? 也叫 RBF核函数、径向基函数
??????? Radial Basic Function Kernel
????????
??????? 则根据下图可知,当 γ 越大的时候,映射的高斯函数就越发陡峭。
????????????????
??????? 在做高斯核映射时,需要选择地标。也就是下图中公式里的 y。
????????
??????? 每个数据点都可以当做地标。
??????? 所以有多少个数据就会映射出多少个维度。
??????? 假定现在有m个维度为n的数据,则高斯核就可以将其映射成mxm维的数据。
??????? 当m非常大的时候,高斯核的运算效率其实很低,但当m并不大甚至小于n的时候,使用核函数就会变得非常高效了。(?存疑 小于)
?
?
?7、非线性SVM代码实现
?
如何选择核函数?
当特征多且接近样本数量,可直接选择线性核SVM。
当特征数少,样本数正常,推荐选用高斯核函数。
当特征数少,样本数很大,建议选择多项式核函数,切记阶数不能过高,否则复杂度过高,难以计算。
?
多项式特征解决非线性问题
import numpy as np
import matplotlib.pyplot as pltfrom sklearn.datasets import make_moons
x, y = make_moons(n_samples=100, noise=0.2, random_state=0)
plt.scatter(x[:,0], x[:,1], c = y)
plt.show()
from sklearn.svm import LinearSVC
lsvc = LinearSVC()
lsvc.fit(x,y)LinearSVC
LinearSVC()
def decision_boundary_plot(X, y, clf):
axis_x1_min, axis_x1_max = X[:,0].min() - 1, X[:,0].max() + 1
axis_x2_min, axis_x2_max = X[:,1].min() - 1, X[:,1].max() + 1
x1, x2 = np.meshgrid( np.arange(axis_x1_min,axis_x1_max, 0.01) , np.arange(axis_x2_min,axis_x2_max, 0.01))
z = clf.predict(np.c_[x1.ravel(),x2.ravel()])
z = z.reshape(x1.shape)
from matplotlib.colors import ListedColormap
custom_cmap = ListedColormap(['#F5B9EF','#BBFFBB','#F9F9CB'])
plt.contourf(x1, x2, z, cmap=custom_cmap)
plt.scatter(X[:,0], X[:,1], c=y)
plt.show()decision_boundary_plot(x,y,lsvc)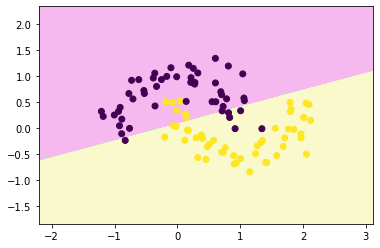
from sklearn.preprocessing import PolynomialFeatures, StandardScaler
from sklearn.pipeline import Pipelinepoly_svc = Pipeline([
("poly", PolynomialFeatures(degree=3)),
("std_scaler", StandardScaler()),
("linearSVC", LinearSVC())
])poly_svc.fit(x,y)Pipeline
Pipeline(steps=[('poly', PolynomialFeatures(degree=3)),
('std_scaler', StandardScaler()), ('linearSVC', LinearSVC())])
PolynomialFeatures
PolynomialFeatures(degree=3)
StandardScaler
StandardScaler()
LinearSVC
LinearSVC()
decision_boundary_plot(x,y,poly_svc)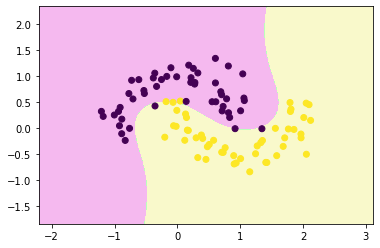
?
核函数解决非线性问题
from sklearn.svm import SVCpoly_svc = Pipeline([
("std_scaler", StandardScaler()),
("polySVC", SVC(kernel='poly', degree=3, coef0=5 ))
])
poly_svc.fit(x,y)Pipeline
Pipeline(steps=[('std_scaler', StandardScaler()),
('polySVC', SVC(coef0=5, kernel='poly'))])
StandardScaler
StandardScaler()
SVC
SVC(coef0=5, kernel='poly')
decision_boundary_plot(x,y,poly_svc)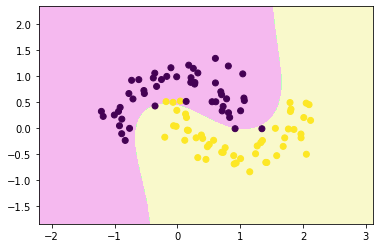
rbf_svc = Pipeline([
("std_scaler", StandardScaler()),
("rbfSVC", SVC(kernel='rbf', gamma=0.1 ))
])
rbf_svc.fit(x,y)
decision_boundary_plot(x,y,rbf_svc)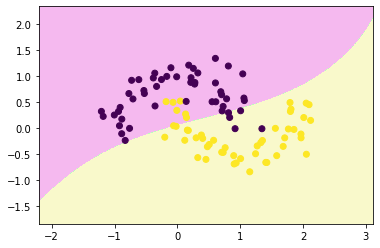
rbf_svc = Pipeline([
("std_scaler", StandardScaler()),
("rbfSVC", SVC(kernel='rbf', gamma=10 ))
])
rbf_svc.fit(x,y)
decision_boundary_plot(x,y,rbf_svc)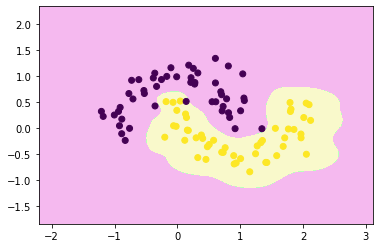
rbf_svc = Pipeline([
("std_scaler", StandardScaler()),
("rbfSVC", SVC(kernel='rbf', gamma=100 ))
])
rbf_svc.fit(x,y)
decision_boundary_plot(x,y,rbf_svc)
?
?
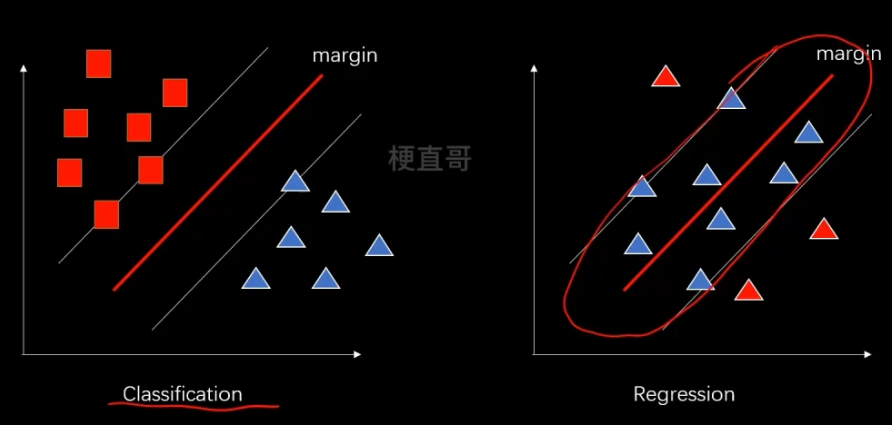
?8、SVM解决回归任务
?
?????????
????????
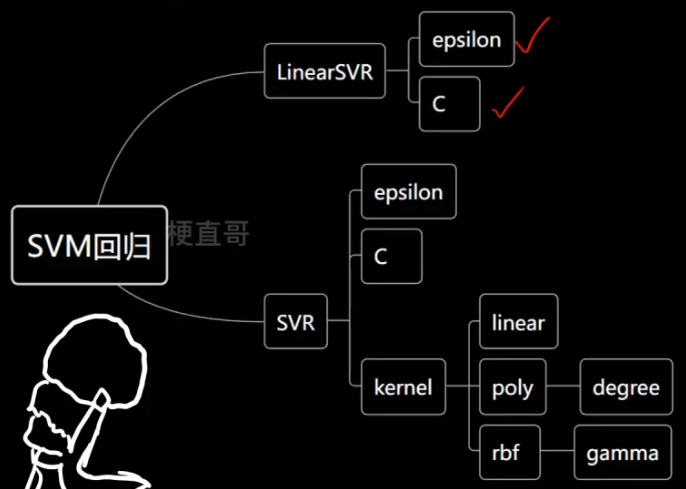
?????????
import numpy as np
import matplotlib.pyplot as pltnp.random.seed(666)
x = np.random.uniform(-3,3,size=100)
y = 0.5 * x**2 +x +2 +np.random.normal(0,1,size=100)
X = x.reshape(-1,1)plt.scatter(x,y)
plt.show()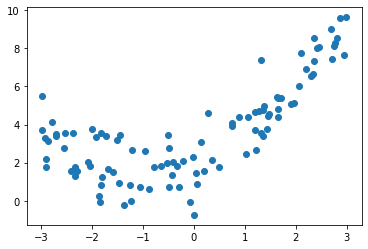
from sklearn.svm import LinearSVR
from sklearn.preprocessing import StandardScaler
from sklearn.pipeline import Pipelinedef StandardLinearSVR(epsilon=0.1):
return Pipeline([
("std_scaler",StandardScaler()),
("linearSVR",LinearSVR(epsilon=epsilon))
])svr = StandardLinearSVR()
svr.fit(X,y)
y_predict = svr.predict(X)
plt.scatter(x,y)
plt.plot(np.sort(x),y_predict[np.argsort(x)],color='r')
plt.show()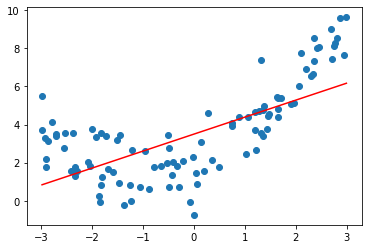
svr.score(X,y)0.48764762540009954
from sklearn.svm import SVRdef StandardSVR(epsilon=0.1):
return Pipeline([
('std_scaler',StandardScaler())
,('SVR',SVR(kernel='rbf',epsilon=epsilon))
])svr = StandardSVR()
svr.fit(X,y)
y_predict = svr.predict(X)
plt.scatter(x,y)
plt.plot(np.sort(x),y_predict[np.argsort(x)],color='r')
plt.show()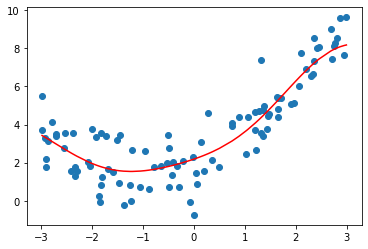
svr.score(X,y)0.8138647789256847
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:veading@qq.com进行投诉反馈,一经查实,立即删除!