基于conda环境下的宏基因组学分析利器MetaWRAP 1.3.2 安装和使用,序列分析基本流程自动分析脚本
介绍:

MetaWRAP是一个宏基因组学分析工具库,用于分析宏基因组测序数据。它提供了一套功能强大的工具,用于进行宏基因组数据的装配、注释和功能分析。
MetaWRAP的功能包括:
-
数据质量控制:包括去除低质量的reads、去除污染序列和剪切适配序列等。
-
基因组装配:MetaWRAP支持多种基因组装配算法,包括SPAdes和MEGAHIT等。根据用户的需求,可以选择不同的算法进行装配。
-
基因组注释:MetaWRAP可以进行基因预测、功能注释和通路预测等操作。它支持使用多种数据库进行注释,包括KEGG、COG和NR等。
-
基因组比较:MetaWRAP可以进行多基因组比较和物种组成分析。它可以帮助用户了解不同样本之间的相似性和差异。
-
生态位分析:MetaWRAP还提供了一些生态位分析工具,可以帮助用户了解样本中微生物的功能和代谢能力。
MetaWRAP的优势包括:
-
丰富的功能:MetaWRAP提供了多种功能,可以帮助用户从原始的测序数据到最终的生物学解释。
-
灵活的使用方式:MetaWRAP支持命令行和Python API两种使用方式,用户可以根据自己的需求选择合适的方式使用。
-
高效的计算性能:MetaWRAP采用了多线程和并行计算的方式,可以加快分析的速度。
总之,MetaWRAP是一个功能强大的宏基因组学分析工具库,可以帮助用户对宏基因组数据进行装配、注释和功能分析。它的使用方式灵活,计算性能高效,适用于各种宏基因组学研究的需要。
凡事先看文章:MetaWRAP—a flexible pipeline for genome-resolved metagenomic data analysis | Microbiome | Full Text
github目录:https://github.com/ursky/metaWRAP
anaconda地址:Login :: Anaconda.org
安装
这里还是介绍conda或mamba安装吧,其他的可能不是最新版,配置起来有时候比较麻烦
conda config --add channels defaults
conda config --add channels conda-forge
conda config --add channels bioconda
conda config --add channels ursky
#
mamba config --add channels defaults
mamba config --add channels conda-forge
mamba config --add channels bioconda
mamba config --add channels ursky安装的时候注意了,直接将所有channel都放上,不然缺包错误:
mamba create -y --name metawrap132 -c ursky -c bioconda -c conda-forge metawrap-mg=1.3.2安装完最后的提示:

查看安装结果,主要看其中metawrap-mg的版本,这里是1.3.2,来自ursky:
mamba list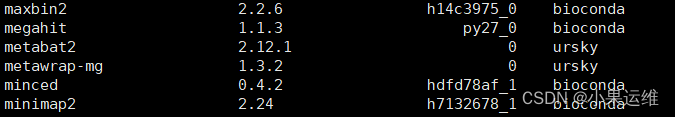
配置建议数据库
这里是各个数据库,数据库大小,以及各个模块可能使用到的数据库,按实际需求配置,如果没有配置对应数据库,则需要在后续模块中指定或着忽略对应参数:

taxonomy数据库:
# 先删除原配置文件夹
rm -rf /miniconda3/envs/metawrap132/opt/krona/taxonomy
# 自己创建指定文件夹
mkdir /path/on/big/disk/taxonomy
# 创建软链接
ln -s /path/on/big/disk/taxonomy /miniconda3/envs/metawrap132/opt/krona/taxonomy
# 自动下载更新数据库,会自动下载到自己指定的文件夹
ktUpdateTaxonomy.sh直接查看ktUpdateTaxonomy.sh文件内容,直接下载来自ncbi数据库:
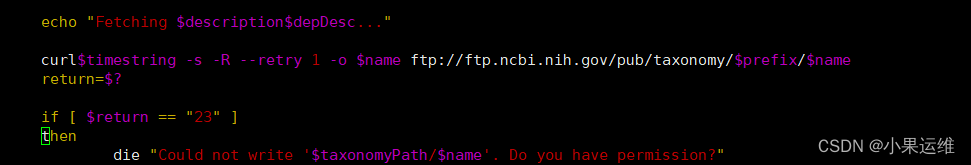

下载解压完成后,在目标目录生成一个taxonomy.tab的文件:
head taxonomy.tab
1 0 1 no rank root
2 2 131567 superkingdom Bacteria
6 7 335928 genus Azorhizobium
7 8 6 species Azorhizobium caulinodans
9 8 32199 species Buchnera aphidicola
10 7 1706371 genus Cellvibrio
11 9 1707 species Cellulomonas gilvus
13 7 203488 genus Dictyoglomus
14 8 13 species Dictyoglomus thermophilum
16 7 32011 genus Methylophilus
GRIDSS\SILVA 16S rRNA\BUSCO数据库
quast-download-gridss
quast-download-silva
quast-download-busco下载后位于目录:
/miniconda3/envs/metawrap/lib/python2.7/site-packages/quast_libs/
下载日志:
envs/metawrap/lib/python2.7/site-packages/quast_libs/silva/blastdb.log
中间几个数据库下载不下来,更换链接吧,应该是版本变了,地址不对:
busco的数据目录:Index of /v5/data/lineages/
busco的官网:BUSCO - from QC to gene prediction and phylogenomics
需要下载的文件:
https://busco-data.ezlab.org/v5/data/lineages/fungi_odb10.2021-06-28.tar.gz
https://busco-data.ezlab.org/v5/data/lineages/eukaryota_odb10.2020-09-10.tar.gz
https://busco-data.ezlab.org/v5/data/lineages/bacteria_odb10.2020-03-06.tar.gz
cd miniconda3/envs/metawrap132/lib/python2.7/site-packages/quast_libs/busco/
mv fungi_odb10.2021-06-28.tar.gz fungi.tar.gz
mv bacteria_odb10.2020-03-06.tar.gz bacteria.tar.gz
mv eukaryota_odb10.2020-09-10.tar.gz eukaryota.tar.gz后面启动quast程序时应该就自动解压了。
主流数据库配置
喜欢原网站信息的个大家可以参考官网:
https://github.com/bxlab/metaWRAP/blob/master/installation/database_installation.md
CheckM
mkdir MY_CHECKM_FOLDER
# Now manually download the database:
cd MY_CHECKM_FOLDER
wget https://data.ace.uq.edu.au/public/CheckM_databases/checkm_data_2015_01_16.tar.gz
tar -xvf *.tar.gz
rm *.gz
cd ../
# Now you need to tell CheckM where to find this data befo本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:veading@qq.com进行投诉反馈,一经查实,立即删除!