大数据HCIE成神之路之数据预处理(1)——缺失值处理
1.1 删除
此方法适用于数据量大、数据缺失少的数据集。使用Python中pandas库的dropna ( ) 函数,其基本格式如下:
DataFrame.dropna(axis=0, how='any', thresh=None, subset=None, inplace=False)
关键参数详解:
- axis=0/1,默认为0。axis=0代表行数据,axis=1代表列数据。
- how=any/all,默认为any。how=any代表若某行或某列中存在缺失值,则删除该行或该列。
- how=all代表若某行或某列中数值全部为空,则删除该行或该列。
- thresh=N,可选参数,代表若某行或某列非缺失值的数量小于N,则删除该行或该列。
- subset=列名,可选参数,代表若指定列中有缺失值,则删除该行。
- inplace=True/False,Boolean数据, 默认为False。inplace=True代表直接对原数据集N做出修改。
- inplace=False代表修改后生成新数据集M,原数据集N保持不变。
1.1.1 实验任务
本课程将介绍基于Python的数据预处理方法,并针对常用预处理方法给出相应的具体编码实现过程。通过学习本课程,学员将掌握基于Python的数据预处理方法,并能够将其运用到实际项目中,解决业务问题。
1.1.1.1 实验背景
利用Python对数据进行删除操作,加深对dropna函数的理解。
1.1.1.2 实验目标
设置dropna函数的各参数删除缺失值。
1.1.1.3 实验数据解析
本实验章节—数据预处理,采用Python自带的iris (鸢尾花)公有数据集进行操作。
鸢尾花数据集是以鸢尾花的特征作为数据来源,数据集包含150个数据记录,分为3类,每类50个数据,每个数据包含4个特征属性和1个目标属性,共5维数据。
4个特征属性的数值为浮点型数据,单位为厘米,分别是:
- Sepal.Length(花萼长度);
- Sepal.Width(花萼宽度);
- Petal.Length(花瓣长度);
- Petal.Width(花瓣宽度))。
1个目标属性为鸢尾花的类别,在数据集中以:0,1,2标识各类别。分别是:
- Iris Setosa(山鸢尾) 为0;
- Iris Versicolour(杂色鸢尾) 为1;
- Iris Virginica(维吉尼亚鸢尾) 为2。
原数据详细信息如下:
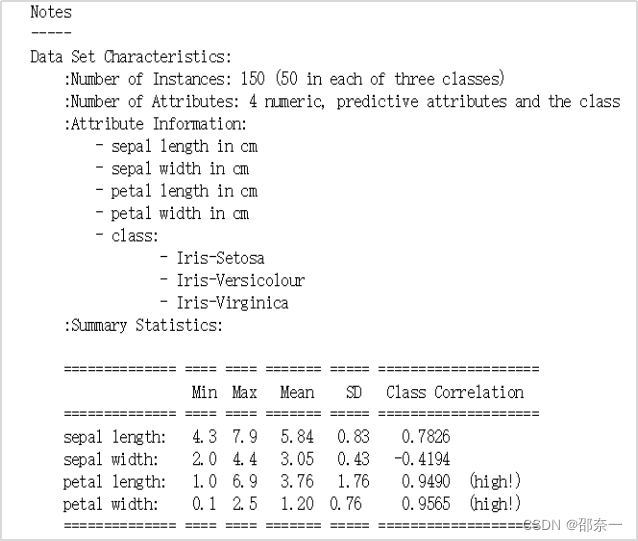
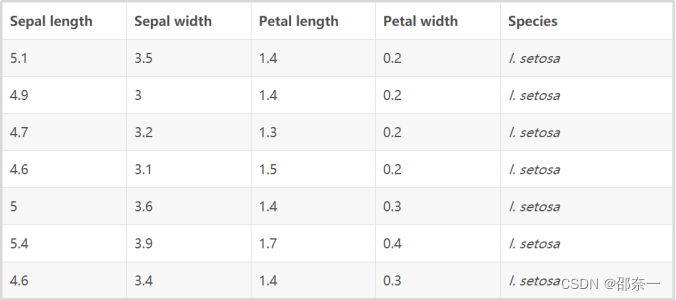
部分原数据可视化后展示:
1.1.2 实验思路
- 导入实验数据集。
- 将数据集转换成实验要求的数据格式。
- 按照删除条件,对数据集进行各类数据删除操作。
1.1.3 实验操作步骤
实验一 删除数据集中的缺失值
步骤 1 数据准备
导入数据集 (大小为150*4),属性列分别为Sepal.Length (花萼长度), Sepal.Width (花萼宽度), Petal.Length (花瓣长度), Petal.Width (花瓣宽度)。
#导入pandas库和numpy库
import pandas as pd
import numpy as np
#导入数据集iris
from sklearn.datasets import load_iris
iris=load_iris()
#部分数据展示
iris
输出结果如下:
{'data': array([[5.1, 3.5, 1.4, 0.2],
[4.9, 3. , 1.4, 0.2],
[4.7, 3.2, 1.3, 0.2],
[4.6, 3.1, 1.5, 0.2],
[5. , 3.6, 1.4, 0.2],
[5.4, 3.9, 1.7, 0.4],
[4.6, 3.4, 1.4, 0.3],
[5. , 3.4, 1.5, 0.2],
[4.4, 2.9, 1.4, 0.2],
说明:第一次执行后需要等待几秒才会出来。
步骤 2 数据转换
dropna() 函数适用于DataFrame结构的数据集,因此需先将iris数据集转换成DataFrame结构,命名为dfx。DataFrame的参数data对应数据集iris.data,columns对应数据属性iris.feature_names。
dfx=pd.DataFrame(data=iris.data, columns=iris.feature_names)
步骤 3 人为加入缺失值
因iris数据集中不含缺失值,因此需人为加入。
#创建名为c的缺失值数据集(名称可自定义),数据集中的属性分别对应于iris中的4个属性
#np.nan为Python中缺失值表示方式
c=pd.DataFrame({"sepal length (cm)":[np.nan,4.9],
"sepal width (cm)":[np.nan,3],
"petal length (cm)":[np.nan,1.4],
"petal width (cm)":[0.2,np.nan]})
c
输出结果如下:
sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 NaN NaN NaN 0.2
1 4.9 3.0 1.4 NaN
使用append函数,将数据集c加入到iris数据集中,形成新数据集df。参数ignore_index为bool型,默认为False。
- ignore_index设置为True。
df=dfx.append(c,ignore_index=True)
df
输出结果如下:
#展示部分加入缺失值的数据集
149 5.9 3.0 5.1 1.8
150 NaN NaN NaN 0.2
151 4.9 3.0 1.4 NaN
152 rows × 4 columns
由上图可看出,新加入了缺失值数据集。因ignore_index=True,所以两组新数据的索引为150和151。
- ignore_index设置为False。
df=dfx.append(c,ignore_index=False)
df
输出结果如下:
#展示部分加入缺失值的数据集
149 5.9 3.0 5.1 1.8
0 NaN NaN NaN 0.2
1 4.9 3.0 1.4 NaN
152 rows × 4 columns
由上图可看出,当ignore_index=False时,新加入数据的索引值保持原形式0和1。
解释:
df=dfx.append(c,ignore_index=True)
#上面这种写法已经过时,不建议使用,可以使用下面这种方式替换:
df=pd.concat([dfx,c],ignore_index=True)
步骤 4 删除缺失值的多种形式展示
已知df数据集中行150和行151含有缺失值 (人为添加),开始进行以下实验:
- 删除含缺失值NaN的数据行。结果如下:
df.dropna()
#关键结果展示
147 6.5 3.0 5.2 2.0
148 6.2 3.4 5.4 2.3
149 5.9 3.0 5.1 1.8
150 rows × 4 columns
df数据集中行150和行151已被删除。
- 删除含缺失值NaN的数据列。结果如下:
df=dfx.append(c,ignore_index=True)
df.dropna(axis='columns')
0
1
2
3
4
5
6
……
因df数据集的4个属性均含有缺失值NaN,因此df数据集完全被删除。
- 删除各属性全为NaN的数据行。因数据集中无数据,则不删除。结果如下:
df=dfx.append(c,ignore_index=True)
df.dropna(how='all')
输出结果如下:
#关键结果展示
149 5.9 3.0 5.1 1.8
150 NaN NaN NaN 0.2
151 4.9 3.0 1.4 NaN
152 rows × 4 columns
- 删除存在缺失值NaN的数据行。结果如下:
df=dfx.append(c,ignore_index=True)
df.dropna(how='any')
输出结果如下:
#关键结果展示
147 6.5 3.0 5.2 2.0
148 6.2 3.4 5.4 2.3
149 5.9 3.0 5.1 1.8
150 rows × 4 columns
删除数据行中非缺失值数量小于2(可自定义)的数据行。
- df数据集中行150数值为[NaN,NaN,NaN,0.2],只有一个数值是非缺失值,缺失值的数量小于2,应删除此数据行。结果如下:
df=dfx.append(c,ignore_index=True)
df.dropna(thresh=2)
输出结果如下:
#关键结果展示
148 6.2 3.4 5.4 2.3
149 5.9 3.0 5.1 1.8
151 4.9 3.0 1.4 NaN
151 rows × 4 columns
删除属性列sepal width (cm)中含有缺失值NaN的数据行。
- df数据集中只有行150中的sepal width (cm)为空,应删除此数据行。结果如下:
df=dfx.append(c,ignore_index=True)
df.dropna(subset=['sepal width (cm)'])
输出结果如下:
#关键结果展示
148 6.2 3.4 5.4 2.3
149 5.9 3.0 5.1 1.8
151 4.9 3.0 1.4 NaN
151 rows × 4 columns
删除含缺失值NaN的数据行,并生成新数据集。结果如下:
df=dfx.append(c,ignore_index=True)
df.dropna(inplace=False)
输出结果如下:
#关键结果展示
147 6.5 3.0 5.2 2.0
148 6.2 3.4 5.4 2.3
149 5.9 3.0 5.1 1.8
150 rows × 4 columns
1.1.4 结果验证
步骤四对数据集进行了多种情况下的删除操作,由各自对应的输出结果可以看出缺失值按照要求删除。
删除缺失值的操作比较多,总结如下方便记忆:
df=dfx.append(c,ignore_index=True)
df=pd.concat([dfx,c],ignore_index=True)
df.dropna(axis='columns')
df.dropna(how='all')
df.dropna(how='any')
df.dropna(thresh=2)
df.dropna(subset=['sepal width (cm)'])
df.dropna(inplace=False)
1.2 填充
填充包括定类-众数填充 (Mode) 和定量-均值、中位数填充 (Mean, Median)。
Python的 sklearn.preprocessing 库中的 Imputer 类可对缺失值进行众数、均值、中位数填充。其基本格式如下:
Imputer(missing_values='NaN', strategy='mean', axis=0, verbose=0, copy=True)
fit_transform() #先拟合数据,再标准化
关键参数详解:
- missing_value,可选参数,int型或NaN,默认为NaN。 这个参数定义了被视为缺失值的实际值。在这里,任何值为 ‘NaN’ 的数据都会被视为缺失值。
- strategy =mean/median/most_frequent,可选参数,定义了用于替换缺失值的策略。
- strategy=mean代表用某行或某列的
均值填充缺失值。 - strategy=median代表用某行或某列的
中位数值填充缺失值。 - strategy =most_frequent代表用某行或某列的
众数填充缺失值。 - axis=0/1,默认为0,可选参数。axis=0代表行数据,axis=1代表列数据。一般实际应用中,axis取0。
- verbose, 默认为0,可选参数。控制Imputer的冗长。
- copy=True/False, Boolean数据, 默认为True,可选参数。copy=True代表创建数据集的副本。
1.2.1 实验任务
1.2.1.1 实验背景
利用Python对数据进行填充操作,加深三种填充方法的理解。
1.2.1.2 实验目标
- 使用均值、中位数、众数填充缺失值。
- 此实验中的数据集沿用实验一中已添加缺失值的数据集df。
1.2.1.3 实验数据解析
实验数据直接使用鸢尾花数据集即可。
1.2.2 实验思路
- 导入实验数据集。
- 对数据集进行均值填充、中位数填充和众数填充。
1.2.3 实验操作步骤
实验二 使用填充方式处理数据集中的缺失值
步骤 1 数据准备
#导入pandas库和numpy库
import pandas as pd
import numpy as np
#导入数据集iris
from sklearn.datasets import load_iris
iris=load_iris()
dfx=pd.DataFrame(data=iris.data, columns=iris.feature_names)
c=pd.DataFrame({"sepal length (cm)":[np.nan,4.9],
"sepal width (cm)":[np.nan,3],
"petal length (cm)":[np.nan,1.4],
"petal width (cm)":[0.2,np.nan]})
#创建学生数据集,包含属性列ID、weight和Height
df=dfx.append(c,ignore_index=True)
df
输出结果如下:
147 6.5 3.0 5.2 2.0
148 6.2 3.4 5.4 2.3
149 5.9 3.0 5.1 1.8
150 NaN NaN NaN 0.2
151 4.9 3.0 1.4 NaN
152 rows × 4 columns
步骤 2 众数填充
以未缺失数据的众数来填充缺失值。红色数据为众数填充结果。
以下的运行结果只是展示,可不执行:
#导入Imputer类
from sklearn.preprocessing import Imputer
#使用Imputer函数进行缺失值填充,参数strategy设置为'most_frequent'代表用众数填充
data = Imputer(missing_values='NaN', strategy='most_frequent', axis=0)
#对填充后的数据做归一化操作
df=dfx.append(c,ignore_index=True)
dataMode = data.fit_transform(df)
#输出dataMode
dataMode
上述代码可能会报错:
ImportError: cannot import name 'Imputer' from 'sklearn.preprocessing'
原因:Imputer类已在Scikit-learn 0.22版本中被弃用,Imputer不在preprocessing里了,而是在sklearn.impute里。而且改名成了SimpleImputer。
解决办法:
第一步: from sklearn.impute import SimpleImputer
第二步:将程序中原来使用的 Imputer 改为 SimpleImputer
第三步: missing_values='NaN' 修改成: missing_values=np.nan
最终实现代码如下:
from sklearn.impute import SimpleImputer
data = SimpleImputer(missing_values=np.nan, strategy='most_frequent')
# 对填充后的数据做归一化操作
df = dfx.append(c, ignore_index=True)
dataMode = data.fit_transform(df)
# 输出 dataMode
dataMode
输出结果如下:
#关键结果展示
[6.5, 3. , 5.2, 2. ],
[6.2, 3.4, 5.4, 2.3],
[5.9, 3. , 5.1, 1.8],
[5. , 3. , 1.5, 0.2],
[4.9, 3. , 1.4, 0.2]])
数据集df经Imputer函数处理后,变成numpy结构。如若想变成DataFrame结构,可进行如下操作。红框部分为众数填充结果。DataFrame内容可参考实验手册“Python语言基础”中的“Pandas基本操作”部分。
mode=pd.DataFrame(data=dataMode,columns=['sepal length (cm)','sepal width (cm)','petal length (cm)','petal width (cm)'])
mode
输出结果如下:
#关键结果展示
sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
149 5.9 3.0 5.1 1.8
150 5.0 3.0 1.5 0.2
151 4.9 3.0 1.4 0.2
步骤 3 均值填充
以未缺失数据的均值来填充缺失值。红色数据为均值填充结果。
旧版本代码如下:
df=dfx.append(c,ignore_index=True)
data = Imputer(missing_values='NaN', strategy='mean', axis=0)
dataMode = data.fit_transform(df)
dataMode
新版本代码如下:
df=dfx.append(c,ignore_index=True)
data = SimpleImputer(missing_values=np.nan, strategy='mean')
dataMode = data.fit_transform(df)
dataMode
输出结果如下:
[6.5 , 3. , 5.2 , 2. ],
[6.2 , 3.4 , 5.4 , 2.3 ],
[5.9 , 3. , 5.1 , 1.8 ],
[5.83708609, 3.05364238, 3.74304636, 0.2 ],
[4.9 , 3. , 1.4 , 1.19205298]]
数据集df经Imputer函数处理后,变成numpy结构。如若想变成DataFrame结构,可按步骤二中的方法转换,这里不再赘述。
步骤 4 中位数填充
以未缺失数据的中位数来填充缺失值。红色数据为中位数填充结果。
旧版本代码如下:
df=dfx.append(c,ignore_index=True)
data = Imputer(missing_values='NaN', strategy='median', axis=0)
dataMode = data.fit_transform(df)
dataMode
新版本代码如下:
df=dfx.append(c,ignore_index=True)
data = SimpleImputer(missing_values=np.nan, strategy='median')
dataMode = data.fit_transform(df)
dataMode
输出结果如下:
[6.5, 3. , 5.2, 2. ],
[6.2, 3.4, 5.4, 2.3],
[5.9, 3. , 5.1, 1.8],
[5.8, 3. , 4.3, 0.2],
[4.9, 3. , 1.4, 1.3]]
1.2.4 结果验证
- 步骤二展示以
众数填充缺失值。经验证,属性值sepal length (cm) 的众数是5.0,属性值sepal width (cm) 的众数是3.0,属性值petal length (cm) 的众数是2.0,属性值petal width (cm) 的众数是0.2。其结果和实验结果一致。 - 步骤三展示以
均值填充缺失值。经验证,属性值sepal length (cm) 的均值是5.83708609,属性值sepal width (cm) 的均值是3.05364238,属性值petal length (cm) 的均值是3.74304636,属性值petal width (cm) 的均值是1.19205298。其结果和实验结果一致。 - 步骤四展示以
中位数填充缺失值。经验证,属性值sepal length (cm) 的中位数是5.8,属性值sepal width (cm) 的中位数是3.0,属性值petal length (cm) 的中位数是4.3,属性值petal width (cm) 的中位数是1.3。其结果和实验结果一致。
1.3 KNN
Python的 sklearn.neighbors 库中的 KNeighborsClassifier 函数可预测缺失值,以便使用预测值填充数据。其基本格式如下:
KNeighborsClassifier(n_neighbors=5, weights='uniform', algorithm='auto', leaf_size=30, p=2, metric='minkowski', metric_params=None, n_jobs=1, **kwargs)
关键参数详解:
n_neighbors可选参数,代表聚类数量,默认为5。weights可选参数,代表近邻的权重值,uniform代表近邻权重一样,distance代表权重为距离的倒数,也可自定义函数确定权重,默认为uniform。algorithm可选参数,代表计算近邻的算法,具体包括{'auto', 'ball_tree', 'kd_tree', 'brute'}。auto会根据传递给fit方法的值自动选择最合适的算法。leaf_size可选参数,这是传递给树的叶节点数量,代表构造树的大小,默认为30。值一般选取默认值即可,太大会影响建模速度。n_jobs可选参数,代表数据计算的jobs数量,选取-1后虽然占据CPU比重会减小,但运行速度会变慢,所有的- core都会运行,默认为1。
可以忽略的参数:
p=2:这是Minkowski距离的幂参数。当p=1时,等同于使用曼哈顿距离,当p=2时,等同于使用欧几里得距离。metric='minkowski':这是用于树的距离度量。'minkowski’表示使用Minkowski距离。metric_params=None:这是度量函数的其他关键字参数。
说明: R语言在KNN填充缺失值方面更为完善和简单,可用 DMwR 包中的
knnImputation(data, k = 10, scale = T, meth = "weighAvg", distData = NULL)
直接对缺失的自变量X进行缺失值填充。可参考如下链接学习:
R语言-缺失值处理1 - 银河统计 - 博客园
KNN法处理缺失数据-Matlab-CSDN博客
1.3.1 实验任务
1.3.1.1 实验背景
利用KNN算法对数据集中的缺失值预测,加深此方法的理解。
1.3.1.2 实验目标
使用KNN预测缺失值Y。测试数据如下,
- “sepal length (cm)”:[5.6,4.9],
- “sepal width (cm)”:[2.5,3],
- “petal length (cm)”:[4.5,1.4],
- “petal width (cm)”:[0.2,2.1]。
1.3.1.3 实验数据解析
实验数据同1.1.1.3。
1.3.2 实验思路
- 导入实验数据集。
- 生成实验测试集。
- 利用实验数据集进行KNN建模。
- 利用建好的KNN模型,对实验测试集中的缺失值进行预测。
1.3.3 实验操作步骤
实验三 使用KNN预测数据集中的缺失值
步骤 1 导入数据集
#导入pandas库和numpy库
import pandas as pd
import numpy as np
#导入数据集iris
from sklearn.datasets import load_iris
iris=load_iris()
# 导入iris数据集,自变量X命名为dfx,分类变量Y命名为dfy
dfx=pd.DataFrame(data=iris.data, columns=iris.feature_names)
dfx
输出结果如下:
# 部分关键数据展示
sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.1 3.5 1.4 0.2
1 4.9 3.0 1.4 0.2
2 4.7 3.2 1.3 0.2
继续打印种类:
dfy=pd.DataFrame(data=iris.target)
dfy
输出结果如下:
# 部分关键数据展示
0
0 0
1 0
2 0
3 0
4 0
... ...
145 2
146 2
147 2
148 2
149 2
步骤 2 生成测试集
因iris无缺失值,因此手动生成不含分类变量的测试集x_test 。
x_test=pd.DataFrame({"sepal length (cm)":[5.6,4.9],
"sepal width (cm)":[2.5,3],
"petal length (cm)":[4.5,1.4],
"petal width (cm)":[0.2,2.1]})
x_test
输出结果如下:
sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.6 2.5 4.5 0.2
1 4.9 3.0 1.4 2.1
步骤 3 建模
基于iris数据集建立KNN模型。
# 导入KNeighborsClassifier
from sklearn.neighbors import KNeighborsClassifier
# KNN模型建立,将此数据集分为三簇,因此n_neighbors=3
modelKNN = KNeighborsClassifier(n_neighbors=3)
modelKNN.fit(dfx,dfy)
步骤 4预测
基于模型modelKNN,使用测试集x_test预测缺失值predicted。
# 预测
predicted = modelKNN.predict(x_test)
# 基于KNN模型,预测缺失值predicted
predicted
输出结果为:
array([1, 0])
基于iris数据集建立的KNN模型预测缺失值Y分别为1和0。
缺失值预测的结果如下:
| 测试集 | 缺失值预测 | |||
| sepal length (cm) | sepal width (cm) | petal length (cm) | petal width (cm) | Y(预测结果) |
| 5.6 | 2.5 | 4.5 | 0.2 | 1 |
| 4.9 | 3 | 1.4 | 2.1 | 0 |
1.3.4 结果验证
经KNN模型预测,测试数据所属类别为1和0。说明在iris数据集中,距离测试数据1最近的点属于类别1,距离测试数据2最近的点属于类别0。
1.4 回归
此方式只适用于缺失值是连续的情况,比如,北京二手房价缺失值填充。因此上述iris数据集不适用于此方法预测缺失值。
Python的 sklearn.linear_model 库中的 LinearRegression 函数可进行回归预测,以便进行数据填充。其基本格式如下:
model = LinearRegression(fit_intercept=True, normalize=False, copy_X=True, n_jobs=1)
关键参数详解:
- fit_intercept=True/Flase,Boolean数据,默认为True,可选参数。fit_intercept =True代表建模是考虑截距。- fit_intercept=Flase代表建模时不考虑截距。
- normalize=True/Flase,Boolean数据, 默认为Flase,可选参数。fit_intercept=Flase代表忽略normalize参数。fit_intercept =True且normalize=True代表回归之前要对自变量X进行归一化。
- copy_X=True/False,Boolean,默认为True,可选参数。copy_X=True代表自变量可以被copy,否则,它将被重写。
- n_jobs,默认为1,可选参数。
思考:“fit_intercept =True且normalize=True代表回归之前要对自变量X进行归一化。”是什么意思?
当fit_intercept=True且normalize=True时,表示在执行线性回归之前,会对自变量X进行归一化处理。归一化是指将数据按比例缩放,使之落入一个小的特定区间。在数学上,这种无量纲化可以在一定程度上消除数据量纲和取值范围的影响,使得不同指标之间具有可比性。
例如,假设我们有以下数据:
X = [[1, 200], [2, 300], [3, 400]]
y = [1, 2, 3]
在这个例子中,X的第二个特征的值远大于第一个特征的值。如果我们直接进行线性回归,可能会导致第二个特征对模型的影响过大。为了解决这个问题,我们可以对X进行归一化处理,使得所有特征的值都在0到1之间。这样,所有特征对模型的影响就相对均衡了。
归一化的公式如下:
X norm = X ? X min X max ? X min X_{\text{norm}} = \frac{X - X_{\text{min}}}{X_{\text{max}} - X_{\text{min}}} Xnorm?=Xmax??Xmin?X?Xmin??
其中, X min X_{\text{min}} Xmin?和 X max X_{\text{max}} Xmax?分别是X的最小值和最大值。
好的,现在可以按照以下步骤对数据进行归一化处理:
-
首先,我们需要找到每个特征的最小值和最大值。在这个例子中,第一个特征的最小值是1,最大值是3;第二个特征的最小值是200,最大值是400。
-
然后,我们将每个特征的值减去该特征的最小值,然后除以该特征的最大值和最小值的差。这样,每个特征的值都会在0到1之间。
具体计算如下:
X_norm = [[(1-1)/(3-1), (200-200)/(400-200)], # 第一个样本
[(2-1)/(3-1), (300-200)/(400-200)], # 第二个样本
[(3-1)/(3-1), (400-200)/(400-200)]] # 第三个样本
所以,归一化后的数据为:
X_norm = [[0.0, 0.0],
[0.5, 0.5],
[1.0, 1.0]]
这样,所有特征的值都在0到1之间,每个特征对模型的影响相对均衡。希望这个解释对你有所帮助!
所以,当fit_intercept=True且normalize=True时,LinearRegression会自动对X进行归一化处理,然后再进行线性回归。这样可以确保所有特征对模型的影响是均衡的。
1.4.1 实验任务
1.4.1.1 实验背景
利用回归算法对数据集中的缺失值预测,加深此方法的理解。
1.4.1.2 实验目标
使用回归模型预测缺失值。假设属性Weight (自变量) 和属性Height(因变量)有关,因此可根据Weight和Height建立回归模型,进而预测缺失值Height。
1.4.1.3 实验数据解析
学生数据集sInfo,属性列为ID、Weight和Height。
回归实验数据如下:
| 序号 | 01 | 02 | 03 | 04 | 05 | 06 | 07 | 08 | 09 | 10 |
|---|---|---|---|---|---|---|---|---|---|---|
| ID | 001 | 002 | 003 | 004 | 005 | 006 | 007 | 008 | 009 | 010 |
| Weight | 55 | 54 | 51 | 45 | 67 | 68 | 67 | 55 | 54 | 56 |
| Height | NA | 165 | NA | 160 | 170 | 168 | 167 | 162 | 169 | 166 |
1.4.2 实验思路
- 导入实验数据集。
- 删除缺失值。
- 利用不含缺失值的实验数据集进行回归建模。
- 基于建好的回归模型,用数据集中含缺失值的数据列预测其中的缺失值。
1.4.3 实验操作步骤
实验四 使用回归预测数据集中的缺失值
步骤 1 导入数据
# 创建学生数据集sInfo,属性列为ID、Weight和Height
import pandas as pd
import numpy as np
sInfo=pd.DataFrame({"ID":['001','002','003','004','005','006','007','008',
'009','010'],"Weight":['55','54','51','45','67','68','67','55','54','56'], "Height":[np.nan,'165',np.nan,'160','170','168','167','162','169','166']})
# 查看数据
sInfo
输出结果如下:
ID Weight Height
0 001 55 NaN
1 002 54 165
2 003 51 NaN
3 004 45 160
4 005 67 170
5 006 68 168
6 007 67 167
7 008 55 162
8 009 54 169
9 010 56 166
观察数据集sInfo可知,sInfo中缺失Height数据。
步骤2 删除缺失值
用 dropna() 删除数据集中的缺失值。
# dropna函数中参数how设置为any,代表删除数据中存在NaN的行数据。参数inplace设置为False,代表生成新的数据集sInfonew (dropna函数的详细见2.2.1.1)。
sInfonew=sInfo.dropna(how='any',inplace=False)
sInfonew
输出结果如下:
ID Weight Height
1 002 54 165
3 004 45 160
4 005 67 170
5 006 68 168
6 007 67 167
7 008 55 162
8 009 54 169
9 010 56 166
可以发现ID为001跟003的这两行身高是空的行已经被删掉了。
步骤3 建立回归模型
基于现有数据建立回归模型,新版本Python要求对数据使用 values.reshape( ) ,确保训练数据维度和模型要求一致。
# 基于两个变量建立回归模型。
from sklearn.linear_model import LinearRegression
model = LinearRegression(copy_X=True, fit_intercept=True, n_jobs=1, normalize=False)
model.fit(sInfonew['Weight'].values.reshape(-1,1),sInfonew['Height'].values. reshape(-1,1))
解释:
model = LinearRegression(copy_X=True, fit_intercept=True, n_jobs=1, normalize=False):这行代码创建了一个LinearRegression对象,并将其赋值给变量model。这个对象将用于拟合线性回归模型。参数的含义如下:copy_X=True:这意味着在进行计算之前,会对输入数据X进行复制。这样可以避免对原始数据进行更改。fit_intercept=True:这意味着模型将包含截距项。如果设置为False,则认为数据已经居中,不会计算截距项。n_jobs=1:这是用于计算的CPU核心数量。1表示只使用一个核心。normalize=False:这意味着在进行回归之前,不会对输入数据X进行归一化处理。如果设置为True,则会对X进行归一化。
model.fit(sInfonew['Weight'].values.reshape(-1,1),sInfonew['Height'].values. reshape(-1,1)):这行代码是用来拟合线性回归模型的。fit方法接受两个参数:X和y,分别代表特征和目标变量。在这个例子中,我们使用Weight作为特征,Height作为目标变量。reshape(-1,1)是用来确保数据的形状正确,-1表示行数由数据的大小决定,1表示列数为1。
思考: n_jobs 此参数应该是多少比较合适?此参数的调整与什么相关?
n_jobs参数决定了在进行模型训练时,计算机用于并行计算的CPU核心数量。这个参数的设定与以下因素相关:
-
计算机的硬件配置:如果你的计算机有多个CPU核心,那么可以设置
n_jobs为一个大于1的整数,以利用多核并行计算的优势,加快模型训练的速度。如果你的计算机只有一个CPU核心,那么n_jobs应该设置为1。 -
模型训练的复杂性:如果你的模型训练任务非常复杂,需要大量的计算资源,那么可以考虑设置
n_jobs为一个较大的值,以提高计算效率。但是,如果你的模型训练任务相对简单,那么即使设置n_jobs为一个较大的值,也可能无法显著提高训练速度。 -
其他任务的需求:如果你的计算机同时还需要处理其他任务,那么可能需要考虑将
n_jobs设置为一个较小的值,以避免占用过多的CPU资源。
值得注意的是,如果你将n_jobs设置为-1,那么sklearn会使用所有可用的CPU核心进行计算。总的来说,n_jobs的设定需要根据你的具体情况进行权衡。
步骤4 基于回归模型预测缺失值
使用构建完成的model预测Height。
# 当Weight为55时,使用模型预测对应Height
y1 = model.predict([[55]])
# y1预测结果
y1
输出结果如下:
array([[164.86855941]])
预测y2对应的Height值。
# 当Weight为51时,使用模型预测对应Height
y2 = model.predict([[51]])
# y2预测结果
y2
输出结果如下:
array([[163.6298633]])
同时预测Weight为55和51的Height值,代码如下:
# 当Weight为55和51时,使用模型预测对应Height
y3 = model.predict([[55],[51]])
# y3预测结果
y3
输出结果如下:
array([[164.86855941],
[163.6298633 ]])
1.4.4 结果验证
根据model预测出当Weight为 55 时,Height为 164.86855941 。当Weight为 51 时,Height为 163.6298633 。
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:veading@qq.com进行投诉反馈,一经查实,立即删除!