【论文阅读笔记】M3Care: Learning with Missing Modalities in Multimodal Healthcare Data
本文介绍了一种名为“M3Care”的模型,旨在处理多模态医疗保健数据中的缺失模态问题。这个模型是端到端的,能够补偿病人缺失模态的信息,以执行临床分析。M3Care不是生成原始缺失数据,而是在潜在空间中估计缺失模态的任务相关信息,利用来自具有相似未缺失模态的其他病人的辅助信息。该模型通过任务引导的模态适应性相似性度量来找到相似的病人,并据此进行临床任务。实验表明,M3Care在多种评估指标上超越了现有的基线模型,并且其发现与专家意见和医学知识一致,显示出提供有用见解的潜力。【开放源码】
一.论文概述
-
端到端模型设计:M3Care是一个端到端的模型,能够直接处理具有缺失模态的患者数据,补偿这些缺失信息,以执行临床分析。
-
任务相关信息的估计:与传统方法不同,M3Care不是直接生成原始缺失数据,而是在潜在空间中估计缺失模态的任务相关信息。这种方法避免了直接处理原始数据的不稳定性和复杂性。
-
利用相似患者的辅助信息:M3Care模型通过任务引导的模态适应性相似性度量来识别相似的患者,并利用这些相似患者的未缺失模态数据来估计目标患者的缺失信息。
-
临床任务的有效执行:模型能够利用估计出的任务相关信息来有效执行临床任务,如疾病诊断或预后预测。
-
实验验证:作者通过在真实世界数据集上的实验,展示了M3Care在各种评估指标上优于现有基线模型的性能,并且其发现与专家意见和医学知识一致,显示出提供有用见解的潜力。
二.模型结构
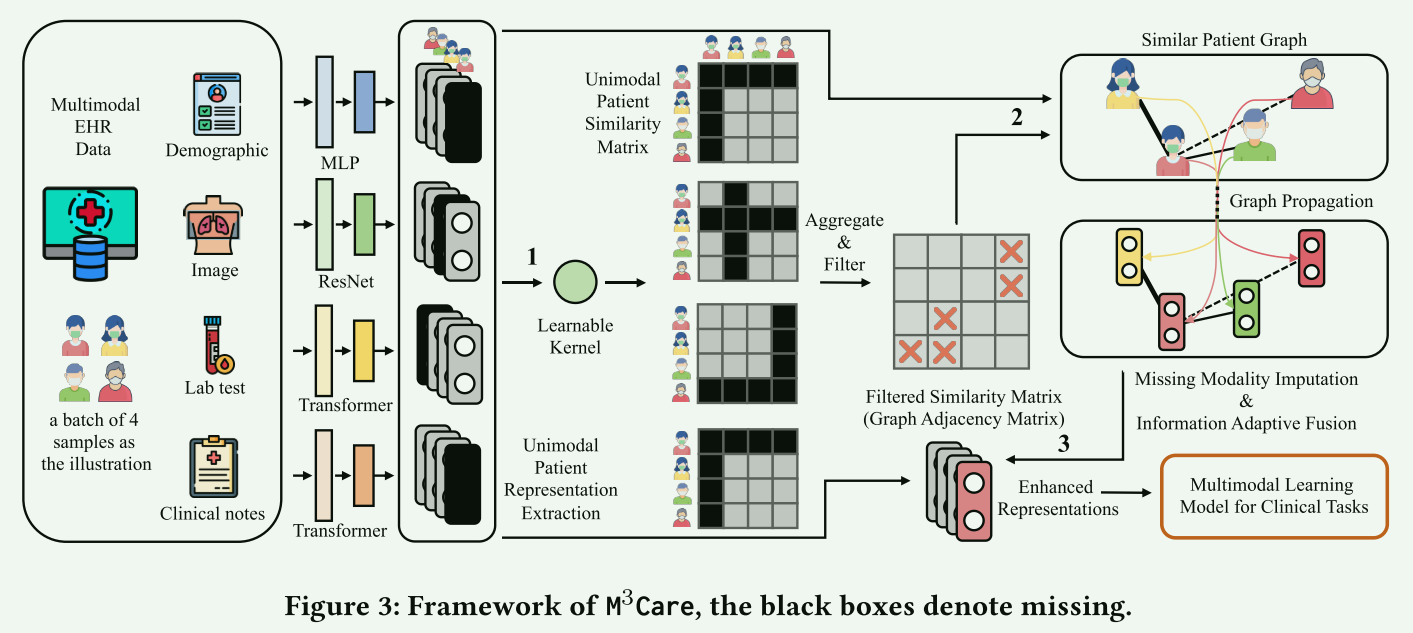

-
Unimodal Representation Extraction:使用不同的特征抽取模型抽取各自模态特征,图上很清楚,不赘述。
-
Similar Patients Discovery and Information Aggregation:包含如下:
-
task-guided modality-semantic-adaptive similarity metric:使用特殊的核函数方法度量,而不是传统的余弦相似度。
k ω m ( ? h i m , ? h j m ) = [ ( 1 ? δ m ) k ( ? ω m ( ? h i m ) , ? ω m ( ? h j m ) ) + δ m ] q ( ? h i m , ? h j m ) k_{\omega_{m}}\left(\mathrm{~h}_{i}^{m}, \mathrm{~h}_{j}^{m}\right)=\left[\left(1-\delta_{m}\right) k\left(\phi_{\omega_{m}}\left(\mathrm{~h}_{i}^{m}\right), \phi_{\omega_{m}}\left(\mathrm{~h}_{j}^{m}\right)\right)+\delta_{m}\right] q\left(\mathrm{~h}_{i}^{m}, \mathrm{~h}_{j}^{m}\right) kωm??(?him?,?hjm?)=[(1?δm?)k(?ωm??(?him?),?ωm??(?hjm?))+δm?]q(?him?,?hjm?)
- 核函数 k ω m ( ? h i m , ? h j m ) k_{\omega_{m}}\left(\mathrm{~h}_{i}^{m}, \mathrm{~h}_{j}^{m}\right) kωm??(?him?,?hjm?):这个函数用于计算两个患者在特定模态 m m m中特征表示的相似度。这里, h i m \mathrm{h}_{i}^{m} him?和 h j m \mathrm{h}_{j}^{m} hjm?分别表示第 i i i和第 j j j 个患者在模态 m m m中的特征表示。
- 混合项:公式中的第一个部分 ( 1 ? δ m ) k ( ? ω m ( ? h i m ) , ? ω m ( ? h j m ) ) \left(1-\delta_{m}\right) k\left(\phi_{\omega_{m}}\left(\mathrm{~h}_{i}^{m}\right), \phi_{\omega_{m}}\left(\mathrm{~h}_{j}^{m}\right)\right) (1?δm?)k(?ωm??(?him?),?ωm??(?hjm?)),其中 ? ω m \phi_{\omega_{m}} ?ωm?? 是一个转换函数,将原始特征空间映射到一个新的空间,以便更好地捕捉相似度。 δ m \delta_{m} δm?是一个模态特定的调整参数,它决定了在核函数中保留多少原始特征空间的信息。
- 权重项 δ m \delta_{m} δm?:这个参数调节着在相似度计算中原始特征空间和转换后空间的相对重要性。它的值在 0 和 1 之间,用于平衡两种不同的相似度计算方式。
- 附加项 q ( ? h i m , ? h j m ) q\left(\mathrm{~h}_{i}^{m}, \mathrm{~h}_{j}^{m}\right) q(?him?,?hjm?):这个项用于在相似度计算中添加额外的信息,可能是基于特定模态 m m m 的特定特征或考虑的其他因素
-
信息聚合:
Π ~ = ∑ 1 M Π m ? mask ? m ∑ 1 M mask ? m + ? Π ~ i , j = { Π ~ i , j ?if? Π ~ i , j > Λ 0 ?if? Π ~ i , j ≤ Λ \begin{array}{c} \tilde{\Pi}=\frac{\sum_{1}^{M} \Pi^{m} \cdot \operatorname{mask}^{m}}{\sum_{1}^{M} \operatorname{mask}^{m}+\epsilon} \\ \tilde{\Pi}_{i, j}=\left\{\begin{array}{cc} \tilde{\Pi}_{i, j} & \text { if } \tilde{\Pi}_{i, j}>\Lambda \\ 0 & \text { if } \tilde{\Pi}_{i, j} \leq \Lambda \end{array}\right. \end{array} Π~=∑1M?maskm+?∑1M?Πm?maskm?Π~i,j?={Π~i,j?0??if?Π~i,j?>Λ?if?Π~i,j?≤Λ??
- Π ~ \tilde{\Pi} Π~:这代表最终聚合后的结果。
- ∑ 1 M Π m ? mask ? m \sum_{1}^{M} \Pi^{m} \cdot \operatorname{mask}^{m} ∑1M?Πm?maskm:这里, Π m \Pi^{m} Πm 表示第 m m m 个模态的某种计算结果或特征表示,而 mask ? m \operatorname{mask}^{m} maskm 是一个掩码(mask),用于指示第 m m m个模态是否可用或重要。掩码通常是二进制的(0或1),用于选择性地考虑(或忽略)特定模态。
- ∑ 1 M mask ? m + ? \sum_{1}^{M} \operatorname{mask}^{m}+\epsilon ∑1M?maskm+?:分母是对所有模态的掩码求和,再加上一个小常数 ? \epsilon ?(通常接近0)以避免除以零的情况。这种求和确保了当某些模态缺失时,计算结果仍然是有意义的。
- Π ~ i , j \tilde{\Pi}_{i, j} Π~i,j?:这是聚合后的结果矩阵中的一个元素,代表第 i个样本和第 j j j个样本之间的某种度量。
- 条件语句:这里的条件语句用于应用一个阈值 Λ \Lambda Λ。如果 Π ~ i , j \tilde{\Pi}_{i, j} Π~i,j?的值大于阈值 Λ \Lambda Λ,它将被保留;如果小于或等于 Λ \Lambda Λ,则将该值设置为0。这种方法用于过滤掉那些低于特定重要性水平的元素。
-
? 总体来说,这个公式描述了一个两步过程:首先是结合多个模态的信息,然后通过应用阈值来过滤和精细化结果。目标是通过合并来自相似患者的辅助信息来推断模态缺失样本。因此,为了聚合来自相似的信息,将一批患者的表示表示为每个模态中的一个图,相似度矩阵 Π ~ \tilde{\Pi} Π~作为图的邻接矩阵(即,然后使用图卷积层(GCN),利用结构信息增强表示学习。
- Adaptive Modality Imputation:
- Multimodal Interaction Capture:这部分没有什么好说,就是常规Transformer融合多模态特征。
三.数据集
Ocular Disease Intelligent Recognition (ODIR) Dataset and Ophthalmic Vitrectomy
(OV) Dataset 眼病智能识别(ODIR)数据集和眼科玻璃体切除术(OV)数据集
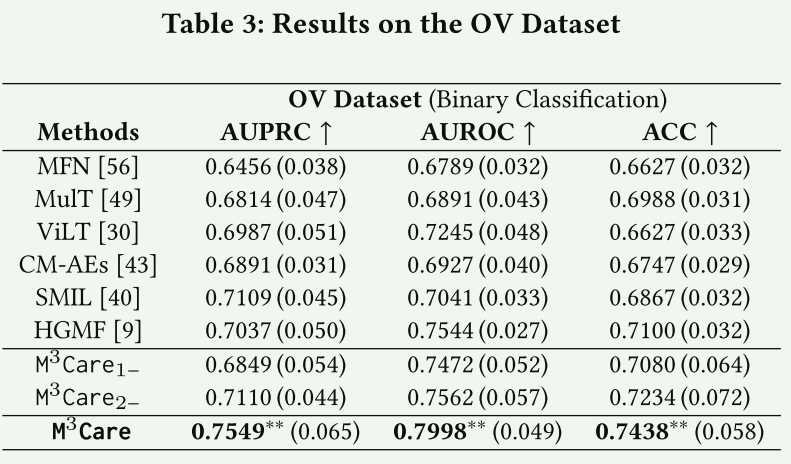
四 .实验结果

本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:veading@qq.com进行投诉反馈,一经查实,立即删除!