Nature Genet|缺氧诱导因子HIF复合体招募组蛋白甲基转移酶SET1B激活缺氧诱导基因
缺氧诱导的转录因子(Hypoxia-inducible transcription factors, HIFs)通过激活大量参与血管生成、pH调节、糖酵解和细胞生长的基因来控制细胞对低氧适应性,从而在缺氧时能够促使细胞存活。除了这种必要的生理反应外,HIFs还参与多种生理过程,包括细胞发育、炎症和免疫调节以及肿瘤生长。因此,深入了解HIFs是如何在低氧环境中如何进行调节的非常重要。
今天跟大家分享一篇来自Nature Genetics的一篇名为The HIF complex recruits the histone methyltransferase SET1B to activate specific hypoxia-inducible genes文章,探究HIF响应低氧过程如何与染色质相互作用。

01 研究背景
HIF调控主要依赖于HIF-α亚基的翻译后氧依赖降解。两种主要的HIF-α亚型(HIF-1α和HIF-2α)在氧气存在时通过脯氨酸羟化酶(PHD)进行脯氨酸羟化,促进Von Hippel-Lindau (VHL) E3连接酶的识别和泛素化,随后的蛋白酶介导其降解。HIF-α的降解使得不能与HIF1β形成HIF异质二聚体转录因子。当氧水平降低,PHD活性降低,则HIF-α稳定,能与HIF1β形成二聚HIF复合物,而后进入细胞核,并与染色质相互作用激活相关基因转录。
虽然HIF蛋白调控机制已经很清楚,但上游靶哪个靶基因调节HIF活性的还不太清楚,有可能是通过染色质可及性和组蛋白尾修饰来控制的。先前研究证实了组蛋修饰的重要性,p300/CBP赖氨酸乙酰转移酶复合物参与了HIF靶基因的表达。组蛋白去甲基化可以通过赖氨酸去甲基化酶(KDM)以氧依赖的方式调控。这些组蛋白修饰标记擦除蛋白与PHD属于相同的酶家族,需要氧气作为共同底物,最近的研究表明,缺氧期间H3K4me3、H3K27me3和H3K36me3水平都会升高。
第一,HIF是否可以招募特定的染色质修饰标记书写者或擦除复合物;第二,组蛋白赖氨酸修饰的形成是否依赖于HIF复合体或针对性在基因启动子区域的存在;作者真对这两个问题进行探究。利用基于流式细胞术的CRISPR/Cas9突变筛查,作者发现SET1B(也称为SETD1B,一种H3K4甲基转移酶),HIF有选择性地招募它,以促进HIF靶基因的快速激活。SET1B调节缺氧诱导的基因对应转录本形成,SET1B缺失会降低缺氧期间癌细胞的存活率,并减少异种移植中的肿瘤形成,并且发现 SET1B的H3K4靶向甲基化与细胞缺氧反应调控有关。
02 技术路线

03 研究结果
1. CRISPR筛选可以识别并激活HIF反应所需的基因
为了确定可激活HIF靶基因位点所需的基因,作者构建了一个动态HIF-mcherry报告系统(图1a-c)。通过构建cas9的全基因组sgRNA文库转导至HeLa细胞中进行目的基因筛选。在1%的氧气中培养细胞与仅暴露于21%氧气的细胞培养24h比较,并对未激活报告细胞的细胞进行流式分选(图1d、e)。再经过高通量测序,分析得到未激活报告基因的分类群体中富集的sgRNAs (图1d, e)。HIF-1β (ARNT)和HIF-2α(EPAS1)在突变筛选中高度富集,结果也说明此筛选模型的可行性(图1e)。结果表明大多数top候选基因涉及DNA结合,还有一些基因注释参与凋亡或细胞信号通路。作者关注的基因在筛选中高度富集,其中包含组蛋白修饰相关蛋白:SET1B, UBE2A (RAD6)和PPP4C(图1e)。
SET1B、UBE2A和PPP4C均在混合CRISPR敲除(KO)的HeLa报告细胞中被筛选到(图1f)。为了探究SET1B介导组蛋白甲基化参与HIF靶基因的激活。作者通过使用混合SET1B基因敲除群体sgRNAs和分离克隆,观察到缺氧时HIF报告基因的激活降低(图1f),在21%的氧气报告基因水平却没有变化(扩展数据图1j),此结果也证实了SET1B的参与细胞缺氧HIF响应。虽然SET1B缺失并不能像HIF1β缺失那样阻止HIF报告因子的活性,但它延迟了HIF的激活降低基因表达水平 (图1g)。
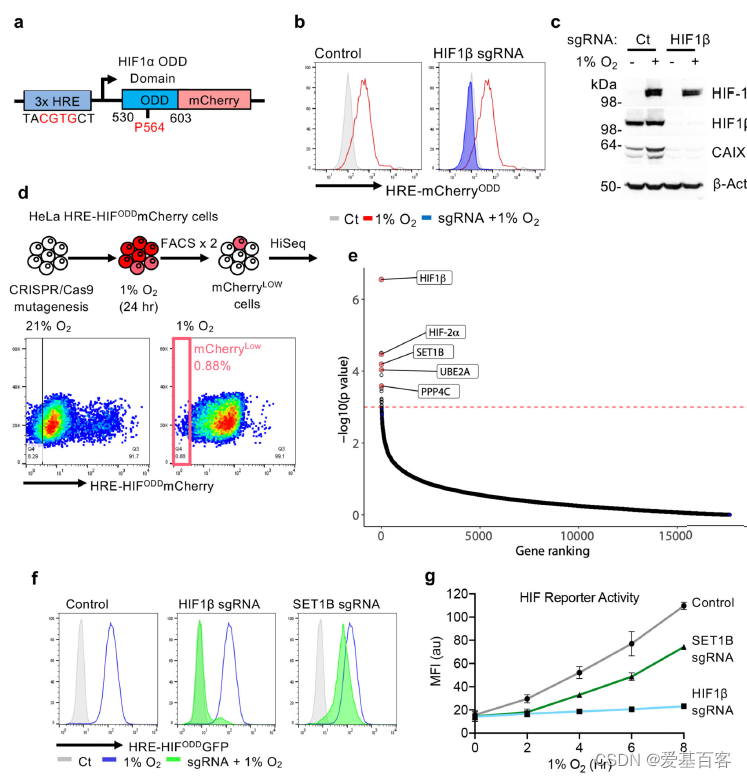
图1 CRISPR筛选可以识别并激活HIF反应所需的基因
2.?SET1B缺失会降低HIF转录反应
为探究SET1B缺失是否改变了内源性HIF靶基因的激活,其他细胞类型是否也有相一致的反应。作者使用经过验证的HIF-1α靶标基因碳酸酐酶9 (CA9)作为检测指标,在HeLa HIF报告中CA9水平下降,在其他癌细胞株(A549肺腺癌和MCF7乳腺癌细胞)和皮肤成纤维细胞(图2c-f)中也观察到了类似的结果。报告基因激活的减少并不是由于HIF-α或HIF1β水平的改变,SET1B缺失并没有改变HIF-1α mRNA表达或HIF-2α或HIF1β蛋白丰度。在siRNA介导的缺失后,对SET1B进行回补,恢复了HIF的活性,并增加了CA9水平(图2g),证实了SET1B参与了HIF相关靶基因的转录激活。
接下来,作者考虑除去缺氧环境,SET1B缺失是否直接可抑制HIF靶基因的激活。利用PHD抑制剂二甲基氧基甘氨酸(DMOG)处理,HIF-1α稳定表达并激活了HIF报告因子。但在SET1B缺失后,HIF报告因子水平和内源性CA9仍然出现下降。此外,蛋白酶体抑制(MG132处理)并不能挽救SET1B缺失后的CA9水平。SET1B增强HIF靶标的转录活性而不是改变HIF-1α的稳定性。
人类已有6种H3K4甲基转移酶被鉴定:SET1A、SET1B和4个混合谱系MLL基因,它们共同组成SET/COMPASS家族。SET1和SET1B密切相关,COMPASS成员复合体除CFP1和WDR82外,还共享多个亚基(图2h)。因此,作者检测了在DMOG处理后,SET1A、CFP1或WDR82的缺失是否改变了HIF报告基因的激活。SET1A缺失对HIF基因的激活没有影响(图2i, j)。WDR82缺失细胞毒性导致没有结果数据,但与SET1A或SET1B相关的CFP1缺失,部分降低了HIF报告基因的激活(图2i, k, l)。这些发现支持SET1B而非SET1A复合体参与HIF反应。

图2 SET1B缺失会抑制HIF的转录
3. SET1B选择性驱动HIF靶基因表达
作者利用RNA-seq检测SET1B对细胞在缺氧时基因表达的整体影响。将SET1B基因敲除、HIF-1β基因敲除、野生型HeLa细胞暴露于21%或1%的氧气中12小时(图3a)。在21%的氧气中,SET1B缺失对整体基因转录的影响很小(图3b, c)。在缺氧中,有1033个基因被显著诱导表达,其中764个(74%)基因在HIF-1β缺失后减少(图3d, e)。SET1B缺失显著降低了约20%缺氧依赖基因的表达(图3d, e),缺氧诱导后的HeLa细胞中(CA9, VEGF和PHD3高度上调(图3e, f)。RT-PCR在几种细胞类型中验证对应结果(图3g)。此外,sirna介导的SET1B缺失后回补可恢复CA9和VEGF的HIF激活。
在多种细胞系中,SET1B缺失并未改变GLUT1的表达,提示一些HIF靶基因可能受到差异调控。SET1B更倾向于改变在21%的氧气中低水平表达的HIF相关基因的上调(图3h)。这种差异效应表明,SET1B可能参与了不同的生物过程。其中,SET1B缺失会优先改变参与血管生成的基因VEGF表达,却对糖酵解的影响较小 (Fig. 3i, j)。

图3 SET1B选择性驱动HIF靶基因表达
4. 缺氧中SET1B缺失的影响
为了进一步探究HIF与SET1B的关联,作者将HeLa、A549细胞进行SET1B敲除,以HIFβ、β2m缺失为对照,检测了缺氧条件下SET1B缺失对细胞生长的影响。HeLa和A549细胞在21%氧气条件下以相似的速度生长。在缺氧24-72小时后,SET1B或HIF β缺失的总细胞数量相较对照减少(图4a, b)。缺氧改变了SET1B自身的水平,在24小时后,HeLa和A549细胞中的SET1B转录和蛋白水平均下降,这说明缺氧降低了SET1B的表达,这是一个负反馈回路。重要的是,SET1A与SET1B缺失不同,即使在21%的氧气条件下,SET1A缺失也会使细胞数量减少,并且HIF靶基因和相关调控基因的mRNA表达都减少,表明其在转录调控中的作用更为全面。
接下来,作者研究了SET1B缺失细胞中细胞数量的减少是否由于增殖减少或细胞死亡增加所致。CFSE染色的细胞示踪显示,HIF1β或SET1B缺失细胞的增殖率没有改变。然而,HIF1β或SET1B缺失后PARP1有所增加,缺氧时细胞凋亡增加。利用SET1B、HIF缺失后建立了HeLa HIF报告细胞的球形培养,并测量圆形度作为球形完整性和凋亡的指标。GFP荧光用来展现球体中心区域缺氧并激活HIF情况。HIF1βKO或联合HIF1/2α缺失完全削弱了这些缺氧中心的HIF报告基因的激活(图4c, d),而SET1B缺失显示GFP激活仅轻度减少(图4c, d),这与之前的研究结果一致(图1f)。在hif缺失的椭球体中,椭球体体积变化很大,但圆度明显下降,椭球体完整性丧失(图4e)。SET1B缺失的球体也表现为圆度缺失(图4e),支持SET1B参与HIF调控的生长和凋亡。
将786-0肾细胞癌异种移植瘤作为探究HIF参与肿瘤形成模型,这些肿瘤高度依赖HIF-2α的形成。成瘤结果显示SET1B和 HIF-1β的缺失不会改变786-0细胞在21%氧气条件下的生长。HIF-1β的缺失完全阻止了肿瘤的形成(图4n, o)。SET1B将肿瘤的生长延迟到一个介于对照组和HIF-1β缺失之间的中间水平,7只小鼠中有3只未能形成肿瘤(图4m, n)。
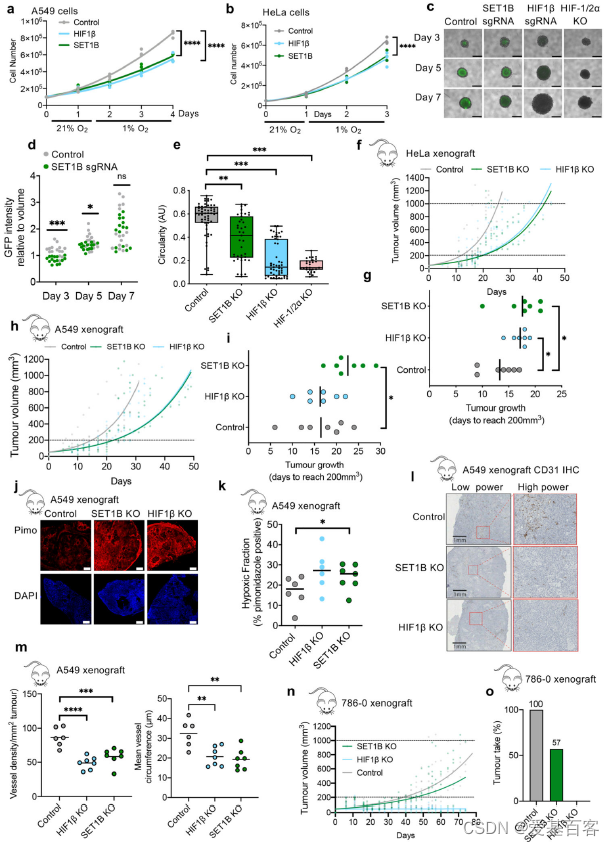
图4 缺氧时SET1B缺失的影响
5. 缺氧时募集SET1B对染色质进行表观重塑
为探究SET1B是如何参与激活HIF靶基因,首先利用COIP检测了SET1B与HIF异源二聚体的相互作用。内源性SET1B与HIF-α亚型和HIF1β亚型相关(图5a)。成熟异源二聚HIF复合物的形成并不是必需的,因为在HIF β缺失细胞中SET1B仍然有HIF-1α富集下来 (图5a)。这种相互作用独立于HIF染色质结合,因为DNase处理并没有破坏HIF-1α与SET1B的结合(图5b)。
为了找到HIF-1α与SET1B复合物的相互作用域,在HEK293T细胞中进行HIF-1α截断突变体,并利用COIP检测了它们与SET1B的结合情况(图5c)。SET1B与包含PAS结构域的HIF-1α相结合,该结构域与HIF-β异质二聚化有关,但不需要ODD或c端反式激活结构域的存在(图5d)。随后的研究证实SET1B与HIF-1α截断结构相关,该结构包含pa-a和pa-b域 (图5e)。因此,SET1B的招募需要HIF-1α上的两个PAS域。
在氧含量为21%的环境下,SET1B定位于胞质和细胞核,提示其不同的定位可能影响其功能。作者提出:缺氧是否改变了SET1B的定位,以及HIF-1α是否将SET1B招募到染色质中。在HeLa和A549细胞的亚细胞定位结果表明,SET1B在21%的氧气中均匀分布在胞质、核质和染色质部分之间,和之前报道的一致,但在缺氧处理后4小时后在染色质部分发现SET1B水平增加。这种SET1B易位与HIF-1α向细胞核的转移密切相关,而SET1A并没有观察到这种易位(图5h)。因此,为了确定HIF复合体是否与SET1B的入核积累有关,检测了HeLa细胞中HIF-1α和HIF-1 β联合缺失后SET1B的定位。缺氧6小时后,SET1B在染色质上积累,但这种增加在HIF-1缺陷细胞中被抑制(图5i)。HIF-2α可能仍部分参与SET1B的招募。
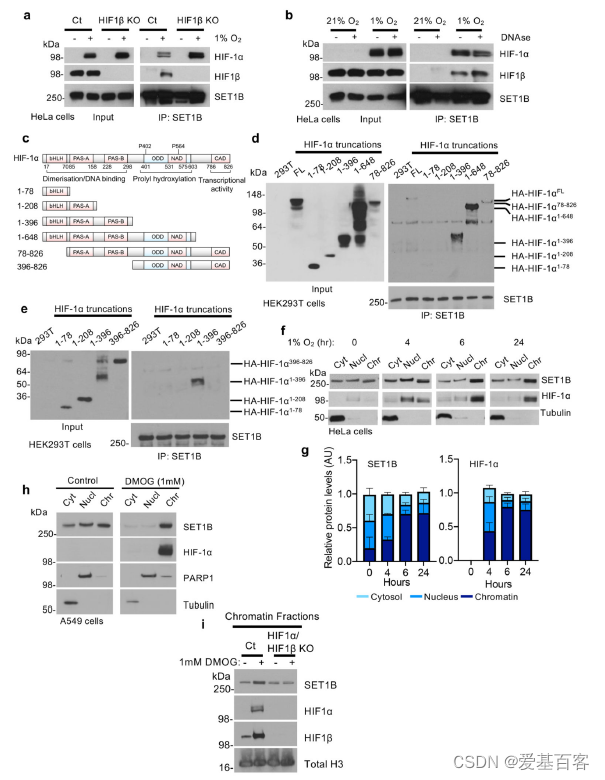
图5 COIP及蛋白截短验证SET1B与HIF结合
为了进一步了解HIF对SET1B的招募作用,利用ChIP-PCR检测HIF靶基因CA9、PHD3,VEGF进行验证,HIF-1a、HIF-1?β的ChIP-qpcr实验结果显示,SET1B只在HIF复合物结合后被招募到HIF靶位点(图6a, b),而在HIF β空细胞中,SET1B或复合物的CFP1成分在HIF-1靶位点上没有结合(图6c,d). SET1B过表达也不能恢复HIF1β缺失细胞中HIF靶基因的激活。因此,虽然SET1B在缺氧期间在细胞核中积累,但它与HIF-1α的关联和招募是需要激活特定的HIF靶基因的表达。

图6 ChIP-qpcr探究HIF下游效应基因的结合
6. 缺氧时依赖SET1B的H3K4me3修饰变化
SET1B是否在缺氧时改变了H3K4me3,以及这是否发生在缺氧或HIF基因位点。暴露于缺氧的HeLa和A549细胞显示H3K4me3总水平出现的瞬时增加,主要归因于缺氧抑制赖氨酸脱甲基酶的活性。H3K4me3在选定的HIF靶点的ChIP-PCR也表明,在缺氧情况下启动子区域的,H3K4me3标记增加(图7a)。然而,H3K4me3增加明显依赖于HIF, HIF β缺失细胞中CA9、PHD3和VEGF启动子区域的H3K4me3水平显著下降,而非HIF靶蛋白BAP1启动子区域的H3K4me3水平没有改变(图7a)。
为了进一步探讨缺氧中SET1B与H3K4甲基化的关系,利用ChIP-seq来确定全基因组H3K4me3水平,并与HIF结合位点(HIF-1α、HIF-2α和HIF-1β ChIP-seq)联合分析。在对照HeLa细胞中,缺氧增加了HIF结合下游的缺氧诱导基因的转录起始位点(TSS)附近H3K4me3水平。在HIF-1β缺失后,这种增加显著减弱。然而,SET1B缺失并没有改变这些HIF靶基因中H3K4me3的总体水平,这与SET1B选择性参与HIF反应相一致。在HIF-1β和SET1B缺失的细胞中,观察到缺氧相关基因的H3K4me3增加(图7b, c)。
通过基因集富集分析(GSEA)将HIF ChIP-seq与RNA-seq分析整合在一起。鉴定到高可信度的HIF结合位点(在6个数据集中至少有4个出现)。相反,在缺氧环境中,这些HIF结合基因在HIF-1β缺失时出现强烈下调。证实了它们对HIF的依赖。有意思的是虽然HIF-1β的缺失降低了TSS和gene body区H3K4me3水平,但SET1B的缺失主要改变了整个gene body的H3K4me3,并且这些基因的H3K4me3的减少在不依赖于hf1 β (图7d)。SET1B KO细胞中H3K4me3缺失从的启动子区向gene body区扩散(图7d)。
最后,作者又探讨了H3K4me3和组蛋白乙酰化之间的关系,正如一些研究强调的启动子甲基化和乙酰化在基因转录控制中的相互作用。通过ChIP-PCR检测了HIF靶基因启动子区域的H3K27ac水平。缺氧时,在特定的HIF靶基因启动子区域H3K27ac增加,但这种H3K27ac的积累被HIF β或SET1B缺失所抑制(图7e),为缺氧时SET1B招募促进基因转录提供了进一步的证据。总之,整个研究结果表明,HIF靶基因的一个子集的组蛋白甲基化是由SET1B介导的,并且HIF通过SET1B复合物的招募来指导这种表观遗传调控,从而揭示了HIF选择性转录调控(图8)。
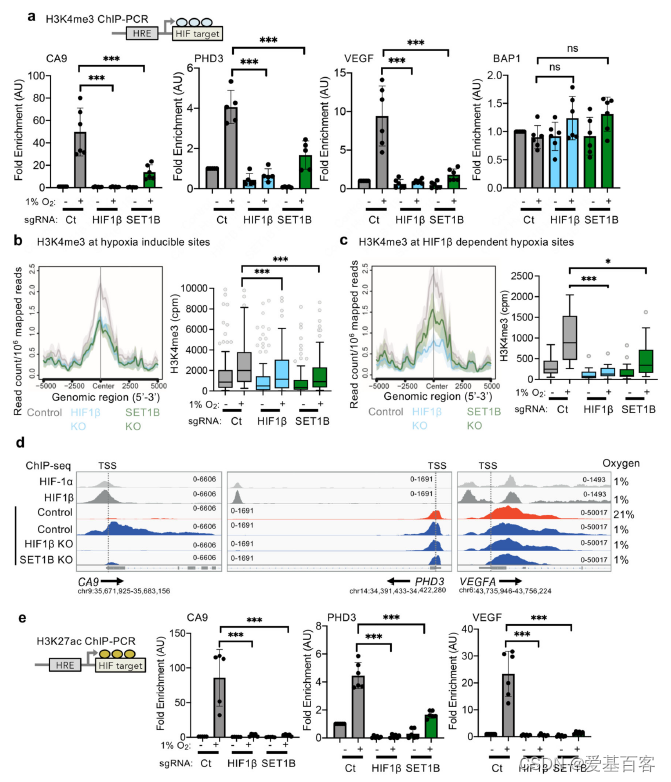
图7 ChIP实验探究 SET1B介导参与组蛋白修饰变化影响缺氧响应基因
04?结? 论?
正常氧环境下,HIF-1α通过氧依赖的蛋白酶体降解,而SET1B主要存在于胞质中。缺氧时,HIF-1α稳定,与HIF-1β形成二聚体,并与SET1B相互作用。缺氧时,SET1B在染色质中积累,HIF复合体引导SET1B到特定的HIF靶基因,在启动子区使得H3K4me3增加。H3K4me3增加可能会招募其他转录调控因子,如乙酰基转移酶(acetyltransferases, Acetyl),促进HIF靶基因表达的激活。

图8 SET1B与HIF在缺氧环境中调控模型
讨论中关于H3K4me3是活性转录的原因还是结果仍存在争议。在本文中并没有针对SET1B可能具有非催化或组蛋白独立功能的可能性进行探究,但数据结果显示,SET1B缺失导致H3K4me3减少,H3K27ac减少,HIF靶基因转录减少,尽管HIF复合物仍然结合在启动子区域。SET1B可能与参与HIF靶基因激活的与乙酰转移酶相互作用,这也将成为下一步研究的重点。组蛋白修饰的动态变化可能也不是由已知的几种酶去进行完全全局性的调整,在不同环境下针对不同基因调控可能也会存在不同;这个过程中一定有很多蛋白参与,还需要科学家们更深入的研究。
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:veading@qq.com进行投诉反馈,一经查实,立即删除!