SAM类医学图像分割选读概述
1.Input augmentation with sam: Boosting medical image segmentation with segmentation foundation model
Zhang Y, Zhou T, Wang S, et al. Input augmentation with sam: Boosting medical image segmentation with segmentation foundation model[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer Nature Switzerland, 2023: 129-139.【开源】
这篇文章想法比较好,既然最近的工作表明,单独使用 SAM,如果没有进一步的微调和域适应,通常无法为医学图像分割任务提供令人满意的结果(在后面几篇文章中都提到了这个观点), 你就将其用于提供更好的先验信息。这里具体解读一下:
【论文概述】
本文提出了一种名为SAMAug的新方法,以提升医学图像分割的效果。这种方法利用了一个称为Segment Anything Model (SAM)的大型基础模型,通过增强医学图像输入来改进常用的医学图像分割模型。尽管SAM在医学图像数据上直接应用时并不立即产生高质量的分割效果,但它生成的掩膜特征和稳定性评分对于构建和训练更好的医学图像分割模型非常有用。文中通过在三个分割任务上的实验,展示了SAMAug方法的有效性。这项工作的主要贡献包括:利用SAM提供的分割输出与原始图像输入的结合,以及为医学图像分割模型构建SAM增强输入图像。此外,还对SAM在医学图像分析中的潜力进行了进一步探索。
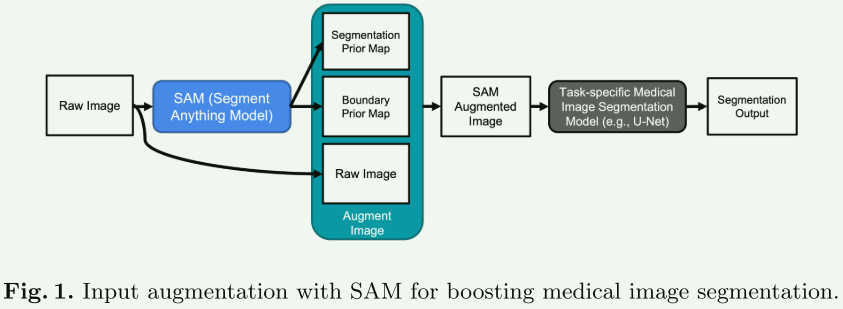
【方法】

- 分割和边界先验图:首先,使用Segment Anything Model (SAM)对医学图像生成分割掩膜。SAM在网格提示设置下为图像的所有可能位置生成分割掩膜。这些掩膜随后被存储在一个列表中,并用于创建分割先验图,同时利用SAM生成的稳定性分数来确定掩膜的值。此外,还生成一个边界先验图,根据SAM提供的掩膜画出每个分割掩膜的外部边界。
- 增强输入图像:将分割先验图添加到原始图像的第二通道,边界先验图添加到第三通道。对于灰度图像,将创建一个三通道图像,包括灰度原始图像、分割先验图和边界先验图。通过这种方式,为训练集中的每个图像生成其增强版本。
- 使用SAM增强的图像进行模型训练:在训练集的每个图像样本上应用输入增强,从而获得一个新的增强训练集。这个增强的训练集用于训练医学图像分割模型。
- 模型部署(测试):在模型部署(测试)阶段,输入同样需要是SAM增强的图像。如果训练模型时同时使用了原始图像和SAM增强图像,就可以在推理时充分发挥训练好的模型的潜力。一种简单的使用方法是对每个测试样本进行两次推理:第一次使用原始图像作为输入,第二次使用SAM增强的图像作为输入。最终的分割输出可以通过两个输出的平均集成来生成。
总的来说,这个方法通过利用SAM生成的先验信息,增强医学图像的输入数据,以改进医学图像分割模型的训练和性能。
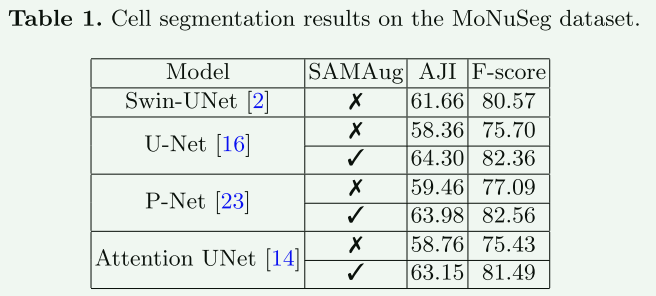
【实验结果】
可以看出经过这种方式处理,涨点 很明显

2. Segment anything model for medical images?
Huang Y, Yang X, Liu L, et al. Segment anything model for medical images?[J]. Medical Image Analysis, 2023: 103061.[【开源】](https://github.com/yuhoo0302/
Segment-Anything-Model-for-Medical-Images.)
本文是直接实验验证,没有改变SAM,不过多解读,只关注结论
为了全面验证SAM在医学数据上的表现,收集并整理了53个开源数据集,并建立了一个大型医学分割数据集,包含18种成像模式、84种物体、125个物体-模式配对目标、1050K 2D图像和6033K掩膜。对所谓的COSMOS 1050K数据集上的不同模型和策略进行了全面分析。发现主要包括
-
SAM在某些特定物体上表现出色,但在其他情况下表现不稳定、不完美甚至完全失败。
-
搭载大型ViT-H的SAM总体性能优于搭载小型ViT-B的。
-
SAM在手动提示(尤其是框提示)下的表现优于"Everything"模式。
-
SAM可以帮助人类进行高质量、节省时间的标注。SAM对中心点和紧凑框提示的随机性敏感,可能会导致严重的性能下降。
-
SAM比只用一个或几个点的交互式方法表现更好,但随着点数增加会被超越。
-
SAM的性能与不同因素(包括边界复杂性、强度差异等)相关。
-
对SAM进行针对特定医学任务的微调可以提高平均DICE性能,分别为ViT-B提高4.39%,ViT-H提高6.68%
3.Segment anything model for medical image analysis: An experimental study
Mazurowski M A, Dong H, Gu H, et al. Segment anything model for medical image analysis: an experimental study[J]. Medical Image Analysis, 2023, 89: 102918.【开源】
本文是直接实验验证,没有改变SAM,不过多解读,只关注结论
SAM的性能取决于数据集和任务的不同而有很大差异,对于某些医学影像数据集表现出色,而对于其他数据集则表现一般或较差。SAM在处理具有明确轮廓和较少模糊性提示的对象(如CT扫描中的器官分割)时表现更好。此外,SAM的性能在使用框提示时比使用点提示时更为突出。尽管SAM在单点提示设置中优于类似方法,但在提供多点提示进行迭代时,SAM的性能通常只有轻微提升,而其他方法的表现则有显著提高。研究还提供了SAM在所有测试数据集上的性能示例,包括迭代分割和在提示模糊情况下的行为。研究结论指出,SAM在特定医学影像数据集上展示了令人印象深刻的零样本分割性能,但在其他数据集上表现适中至较差,其在自动化医学影像分割中具有重要潜力,但在使用时需要谨慎。
4.Segment Anything in Medical Images
Ma J, Wang B. Segment anything in medical images[J]. arXiv preprint arXiv:2304.12306, 2023.【开源】
本文是直接实验验证,没有改变SAM,不过多解读,只关注结论和局限性。
贡献:本文介绍了MedSAM(医学图像分割的基础模型),这是一种为医学图像分割设计的创新工具。MedSAM利用了超过一百万张医学图像构建的大规模数据集,能够在多种分割任务中显示出卓越的性能。与传统的专门化模型相比,MedSAM不仅展示了更好的通用性,而且在某些情况下甚至超越了这些模型。MedSAM的关键特点包括能够处理各种解剖结构、病理条件和医学成像方式,以及对用户提示(如边界框)的响应能力,从而实现精确的目标区域分割。
局限性:训练集中的模态不平衡,其中 CT、MRI和内窥镜图像在数据集中占主导地位。这可能会影响模型在较少代表性的模式(例如乳房X光检查)上的性能。另一个限制是它在分割血管状分支结构方面存在困难,因为在此设置中边界框提示可能不明确。
5.Medical sam adapter: Adapting segment anything model for medical image segmentation
Wu J, Fu R, Fang H, et al. Medical sam adapter: Adapting segment anything model for medical image segmentation[J]. arXiv preprint arXiv:2304.12620, 2023.【开源】
本文同样不直接在医学数据集上微调,而是通过适配器改进。
【论文概述】
这篇论文的核心思想是开发和验证一种名为“医学SAM适配器”(Medical SAM Adapter, MSA)的新技术,用于提升医学图像分割的效能。作者们指出,虽然“分割任何事物模型”(Segment Anything Model, SAM)在图像分割领域表现出色,但在医学图像分割方面却表现不佳。为了解决这个问题,他们提出了一种简单但有效的适配技术,通过整合特定于医学领域的知识到分割模型中。MSA展示了在19种医学图像分割任务上的卓越性能,包括CT、MRI、超声波图像、眼底图像和皮肤镜图像等多种图像模态。这项工作不仅展示了使用参数高效的适配技术可以显著提高原始SAM模型的性能,而且还表明将强大的通用分割模型转移到医学应用领域是可行的。
【方法】

- SAM架构的概述:SAM模型包括三个主要组件:图像编码器、提示编码器和掩码解码器。图像编码器基于标准的视觉变换器(Vision Transformer, ViT),由掩码自编码器(MAE)预训练。此外,论文专注于稀疏编码器,将点和框表示为位置编码,并与每种提示类型的学习嵌入相加。
- 适配器的部署:在SAM编码器的每个ViT块中部署了两个适配器(bottleneck结构:down-projection, ReLU activation, and up-projection)。第一个适配器位于多头注意力之后和残差连接之前,第二个适配器位于多头注意力后的MLP层的残差路径中。在SAM解码器中,每个ViT块部署了三个适配器。没有完全改变所有参数,而是冻结预训练的 SAM 参数并插入适配器模块进行微调。
- 训练策略:作者使用医学图像对模型编码器进行预训练,包括四个医学图像数据集:RadImageNet(包含1.35百万放射影像),EyePACSp(包含88702张彩色眼底图像),BCN-20000和HAM-10000(共约30000张皮肤镜图像)。
- 自监督学习方法:与SAM使用的MAE预训练不同,本文采用了多种自监督学习方法进行预训练,包括对比嵌入混合(Contrastive Embedding-Mixup, e-Mix)和打乱嵌入预测(Shuffled Embedding Prediction, ShED)。
- 针对医学图像的调整:虽然SAM在自然图像上表现出色,但在医学图像分割上表现不佳,主要原因是缺乏针对医学应用的训练数据。为此,本文尝试以最小的努力将SAM扩展到基于提示的医学图像分割。
6.Towards Segment Anything Model (SAM) for Medical Image Segmentation: A Survey
Zhang Y, Jiao R. Towards Segment Anything Model (SAM) for Medical Image Segmentation: A Survey[J]. arXiv preprint arXiv:2305.03678, 2023.【开源】
【文章概述】
尽管SAM在自然图像分割中表现出色,但由于医学图像与自然图像在结构复杂度、对比度和观察者间可变性方面的显著差异,SAM在医学图像分割中的适用性尚不清楚。这篇综述文章提供了对SAM在医学图像分割任务中应用的全面了解,包括其现有的性能、挑战、改进方向以及未来的发展潜力。
【论文中提到的将SAM应用的医学领域的几种方式】
-
提高对不同提示的鲁棒性
由于SAM直接应用于医学图像分割的性能不令人满意,许多研究集中于微调SAM的一小部分参数,如图像编码器、提示编码器和掩码解码器。这种微调的目的是提高SAM在特定医学图像分割任务上的可靠性和效果。
-
将 SAM 的可用性扩展到医学图像
医学图像通常有特定的格式,如NII和DICOM。为了简化SAM在这些格式上的使用,一些研究将SAM集成到常用的医学图像查看器中,例如3D Slicer,从而使研究人员能够在0.6秒的延迟内对医学图像进行分割。这种集成使得SAM能够自动地应用于连续的切片,从而提高其在医学图像处理中的实用性。
-
提高对不同提示的鲁棒性
尽管在医学数据集上微调SAM可以提高性能,但它仍然需要使用手动给出的框或点,这使得实现完全自动的医学图像分割变得困难。此外,最终的分割结果高度依赖于输入的提示,而且模型对错误的提示更为敏感。这表明需要进一步的方法来提高SAM对不同提示的鲁棒性。
-
利用SAM进行输入增强
由于SAM在直接应用于需要领域特定知识的医学图像分割任务时表现不佳,直接利用SAM生成的分割掩码来增强原始的医学图像输入。这种输入增强是通过融合功能实现的,目的是利用SAM生成的分割掩码来改善原始医学图像的分割性能。
【未来的发展】
- SAM在医学图像分割的应用: 直接应用SAM于医学图像分割时,其性能在不同数据集和任务中表现出显著差异。SAM在处理具有不规则形状、弱边界、小尺寸或低对比度的对象时可能会产生不良结果甚至完全失败。对于大多数情况,SAM的分割性能不足以满足特别是在医学图像分割任务中对极高精度的需求。研究表明,适当的微调策略可以在一定程度上改善SAM在医学图像上的分割结果,使其与领域特定模型相媲美。
- 建立大规模医学数据集: 由于自然图像与医学图像之间的显著差异,构建大规模医学数据集对于开发通用的医学图像分割模型至关重要。这将有助于SAM等基础模型在医学图像分割领域的未来发展。
- 整合临床领域知识: 通过整合与图像信息之外的临床信息,如肿瘤位置、大小和预期效果,可以提高SAM的医学图像分割能力。
- 将SAM从2D适应到3D医学图像: 许多医学扫描如MRI和CT是3D体积,这与经典的2D自然图像不同。虽然SAM可以应用于3D医学图像的每个切片,但忽略了相邻切片之间的信息。已有研究表明,相邻切片之间的相关性对于某些对象的识别至关重要。
- 降低医学图像分割的标注成本: 医学图像的高标注成本是开发分割模型的主要挑战之一。尽管SAM生成的分割结果并不总是完美的,但这些分割掩码可以用来减少标注成本。专家可以利用SAM进行粗略分割,然后手动修正以实现快速交互式分割。
- 将SAM集成到临床工作流中: 研究人员可以探索将SAM与眼动追踪技术等结合,设计协作式人机交互系统,以便放射科医师通过简单地观察感兴趣的区域来获取分割掩码。此外,SAM也可以用于提供初步分割,并筛选出需要临床专家更深入审查的复杂病例,从而减轻临床专家的负担。
总体来说,尽管SAM目前与领域特定模型相比性能不稳定,但它在建立医学图像分割基础模型方面展现了巨大的潜力,并有望作为一个高效且强大的工具,进一步协助临床应用。
【文章的彩蛋】
这篇文章的宝藏是其提供了一个不断更新的仓库,罗列了设计到的SAM应用于医学图像方面的论文和数据集。这里把仓库中的内容复制了一下,截至2023.12月:
- Large-scale Datasets for Foundation Models for Medical Imaging.
| Date | Authors | Title | Dataset |
|---|---|---|---|
| 202311 | J. Ye et al. | SA-Med2D-20M Dataset: Segment Anything in 2D Medical Imaging with 20 Million masks (paper) | Link |
- Literature Reviews of Applying SAM for Medical Image Segmentation.
| Date | Authors | Title | Code |
|---|---|---|---|
| 202312 | W. Yue et al. | Part to Whole: Collaborative Prompting for Surgical Instrument Segmentation (paper) | Code |
| 202312 | ZM. Colbert et al. | Repurposing Traditional U-Net Predictions for Sparse SAM Prompting in Medical Image Segmentation (paper) | None |
| 202312 | W. Xie et al. | SAM Fewshot Finetuning for Anatomical Segmentation in Medical Images (paper) | None |
| 202312 | JG. Almeida et al. | Testing the Segment Anything Model on radiology data (paper) | None |
| 202312 | M. Barakat et al. | Towards SAMBA: Segment Anything Model for Brain Tumor Segmentation in Sub-Sharan African Populations (paper) | None |
| 202312 | Y. Zhang et al. | SQA-SAM: Segmentation Quality Assessment for Medical Images Utilizing the Segment Anything Model (paper) | Code |
| 202312 | S. Chen et al. | ASLseg: Adapting SAM in the Loop for Semi-supervised Liver Tumor Segmentation (paper) | None |
| 202312 | HE. Wong et al. | ScribblePrompt: Fast and Flexible Interactive Segmentation for Any Medical Image (paper) | Code |
| 202312 | Y. Zhang et al. | SemiSAM: Exploring SAM for Enhancing Semi-Supervised Medical Image Segmentation with Extremely Limited Annotations (paper) | None |
| 202312 | Y. Zhao et al. | Segment Anything Model-guided Collaborative Learning Network for Scribble-supervised Polyp Segmentation (paper) | None |
| 202311 | N. Li et al. | Segment Anything Model for Semi-Supervised Medical Image Segmentation via Selecting Reliable Pseudo-Labels (paper) | None |
| 202311 | X. Wei et al. | I-MedSAM: Implicit Medical Image Segmentation with Segment Anything (paper) | None |
| 202311 | Z. Shui et al. | Unleashing the Power of Prompt-driven Nucleus Instance Segmentation (paper) | Code |
| 202311 | M. Li and G. Yang et al. | Where to Begin? From Random to Foundation Model Instructed Initialization in Federated Learning for Medical Image Segmentation (paper) | None |
| 202311 | AK. Tyagi et al. | Guided Prompting in SAM for Weakly Supervised Cell Segmentation in Histopathological Images (paper) | Code |
| 202311 | Y. Du et al. | SegVol: Universal and Interactive Volumetric Medical Image Segmentation (paper) | Code |
| 202311 | DM. Nguyen et al. | On the Out of Distribution Robustness of Foundation Models in Medical Image Segmentation (paper) | None |
| 202311 | U. Israel et al. | A Foundation Model for Cell Segmentation (paper) | Code |
| 202311 | Q. Quan et al. | Slide-SAM: Medical SAM Meets Sliding Window (paper) | None |
| 202311 | Y. Zhang et al. | Segment Anything Model with Uncertainty Rectification for Auto-Prompting Medical Image Segmentation (paper) | Code |
| 202311 | Y. Wang et al. | SAMIHS: Adaptation of Segment Anything Model for Intracranial Hemorrhage Segmentation (paper) | Code |
| 202311 | H. Jiang et al. | GlanceSeg: Real-time microangioma lesion segmentation with gaze map-guided foundation model for early detection of diabetic retinopathy (paper) | None |
| 202311 | Y. Xu et al. | EviPrompt: A Training-Free Evidential Prompt Generation Method for Segment Anything Model in Medical Images (paper) | None |
| 202311 | DL. Ferreira and R. Arnaout | Are foundation models efficient for medical image segmentation? (paper) | Code |
| 202310 | H. Li et al. | Promise:Prompt-driven 3D Medical Image Segmentation Using Pretrained Image Foundation Models (paper) | Code |
| 202310 | D. Anand et al. | One-shot Localization and Segmentation of Medical Images with Foundation Models (paper) | None |
| 202310 | H. Wang et al. | SAM-Med3D (paper) | Code |
| 202310 | SK. Kim et al. | Evaluation and improvement of Segment Anything Model for interactive histopathology image segmentation (paper) | Code |
| 202310 | X. Chen et al. | SAM-OCTA: Prompting Segment-Anything for OCTA Image Segmentation (paper) | Code |
| 202310 | M. Peivandi et al. | Empirical Evaluation of the Segment Anything Model (SAM) for Brain Tumor Segmentation (paper) | None |
| 202310 | H. Ravishankar et al. | SonoSAM - Segment Anything on Ultrasound Images (paper) | None |
| 202310 | A. Ranem et al. | Exploring SAM Ablations for Enhancing Medical Segmentation in Radiology and Pathology (paper) | None |
| 202310 | S. Pandey et al. | Comprehensive Multimodal Segmentation in Medical Imaging: Combining YOLOv8 with SAM and HQ-SAM Models (paper) | None |
| 202309 | Y. Li et al. | nnSAM: Plug-and-play Segment Anything Model Improves nnUNet Performance (paper) | Code |
| 202309 | Y. Zhao et al. | MFS Enhanced SAM: Achieving Superior Performance in Bimodal Few-shot Segmentation (paper) | Code |
| 202309 | C. Wang et al. | SAM-OCTA: A Fine-Tuning Strategy for Applying Foundation Model to OCTA Image Segmentation Tasks (paper) | Code |
| 202309 | Y. Zhang et al. | 3D-U-SAM Network For Few-shot Tooth Segmentation in CBCT Images (paper) | None |
| 202309 | CJ. Chao et al. | Comparative Eminence: Foundation versus Domain-Specific Model for Cardiac Ultrasound Segmentation (paper) | None |
| 202309 | H. Ning et al. | An Accurate and Efficient Neural Network for OCTA Vessel Segmentation and a New Dataset (paper) | Code |
| 202309 | C. Chen et al. | MA-SAM: Modality-agnostic SAM Adaptation for 3D Medical Image Segmentation (paper) | Code |
| 202309 | P. Zhang and Y. Wang | Segment Anything Model for Brain Tumor Segmentation (paper) | None |
| 202309 | B. Fazekas et al. | Adapting Segment Anything Model (SAM) for Retinal OCT (paper) | None |
| 202309 | X. Lin et al. | SAMUS: Adapting Segment Anything Model for Clinically-Friendly and Generalizable Ultrasound Image Segmentation (paper) | Code |
| 202309 | X. Xing et al. | SegmentAnything helps microscopy images based automatic and quantitative organoid detection and analysis (paper) | Code |
| 202309 | NT. Bui et al. | SAM3D: Segment Anything Model in Volumetric Medical Images (paper) | Code |
| 202308 | Y. Zhang et al. | Self-Sampling Meta SAM: Enhancing Few-shot Medical Image Segmentation with Meta-Learning (paper) | None |
| 202308 | J. Cheng et al. | SAM-Med2D (paper) | Code |
| 202308 | C. Li et al. | Auto-Prompting SAM for Mobile Friendly 3D Medical Image Segmentation (paper) | None |
| 202308 | W. Feng et al. | Cheap Lunch for Medical Image Segmentation by Fine-tuning SAM on Few Exemplars (paper) | None |
| 202308 | Y. Zhang et al. | SamDSK: Combining Segment Anything Model with Domain-Specific Knowledge for Semi-Supervised Learning in Medical Image Segmentation (paper) | None |
| 202308 | A. Lou et al. | SAMSNeRF: Segment Anything Model (SAM) Guides Dynamic Surgical Scene Reconstruction by Neural Radiance Field (NeRF) (paper) | Code |
| 202308 | A. Archit et al. | Segment Anything for Microscopy (paper) | Code |
| 202308 | X. Yao et al. | False Negative/Positive Control for SAM on Noisy Medical Images (paper) | Code |
| 202308 | B. Fazekas et al. | SAMedOCT: Adapting Segment Anything Model (SAM) for Retinal OCT (paper) | None |
| 202308 | W. Yue et al. | SurgicalSAM: Efficient Class Promptable Surgical Instrument Segmentation (paper) | Code |
| 202308 | H. Zhang et al. | CARE: A Large Scale CT Image Dataset and Clinical Applicable Benchmark Model for Rectal Cancer Segmentation (paper) | Code |
| 202308 | Q. Wu et al. | Self-Prompting Large Vision Models for Few-Shot Medical Image Segmentation (paper) | Code |
| 202308 | A. Wang et al. | SAM Meets Robotic Surgery: An Empirical Study on Generalization, Robustness and Adaptation (paper) | None |
| 202308 | D. Shin et al. | CEmb-SAM: Segment Anything Model with Condition Embedding for Joint Learning from Heterogeneous Datasets (paper) | None |
| 202308 | R. Biswas | Polyp-SAM++: Can A Text Guided SAM Perform Better for Polyp Segmentation? (paper) | Code |
| 202308 | S. Cao et al. | TongueSAM: An Universal Tongue Segmentation Model Based on SAM with Zero-Shot (paper) | Code |
| 202308 | X. Li et al. | Leverage Weakly Annotation to Pixel-wise Annotation via Zero-shot Segment Anything Model for Molecular-empowered Learning (paper) | None |
| 202308 | JN. Paranjape et al. | AdaptiveSAM: Towards Efficient Tuning of SAM for Surgical Scene Segmentation (paper) | Code |
| 202308 | Z. Huang et al. | Push the Boundary of SAM: A Pseudo-label Correction Framework for Medical Segmentation (paper) | None |
| 202307 | J. Zhang et al. | SAM-Path: A Segment Anything Model for Semantic Segmentation in Digital Pathology (paper) | None |
| 202307 | MS. Hossain et al. | Robust HER2 Grading of Breast Cancer Patients using Zero-shot Segment Anything Model (SAM) (paper) | None |
| 202307 | C. Wang et al. | SAMMed : A medical image annotation framework based on large vision model (paper) | None |
| 202307 | G. Deng et al. | SAM-U: Multi-box prompts triggered uncertainty estimation for reliable SAM in medical image (paper) | None |
| 202307 | H. Kim et al. | Empirical Analysis of a Segmentation Foundation Model in Prostate Imaging (paper) | None |
| 202307 | X. Shi et al. | Cross-modality Attention Adapter: A Glioma Segmentation Fine-tuning Method for SAM Using Multimodal Brain MR Images (paper) | None |
| 202307 | C. Cui et al. | All-in-SAM: from Weak Annotation to Pixel-wise Nuclei Segmentation with Prompt-based Finetuning (paper) | None |
| 202306 | E. Kellener et al. | Utilizing Segment Anything Model for Assessing Localization of Grad-CAM in Medical Imaging (paper) | None |
| 202306 | F. H?rst et al. | CellViT: Vision Transformers for Precise Cell Segmentation and Classification (paper) | Code |
| 202306 | W. Lei et al. | MedLSAM: Localize and Segment Anything Model for 3D Medical Images (paper) | Code |
| 202306 | X. Hu et al. | How to Efficiently Adapt Large Segmentation Model (SAM) to Medical Images (paper) | Code |
| 202306 | S. Gong et al. | 3DSAM-adapter: Holistic Adaptation of SAM from 2D to 3D for Promptable Medical Image Segmentation (paper) | Code |
| 202306 | DMH. Nguyen et al. | LVM-Med: Learning Large-Scale Self-Supervised Vision Models for Medical Imaging via Second-order Graph Matching (paper) | Code |
| 202306 | S. Chai et al. | Ladder Fine-tuning approach for SAM integrating complementary network (paper) | Code |
| 202306 | L. Zhang et al. | Segment Anything Model (SAM) for Radiation Oncology (paper) | None |
| 202306 | G. Ning et al. | The potential of ‘Segment Anything’ (SAM) for universal intelligent ultrasound image guidance (paper) | None |
| 202306 | C. Shen et al. | Temporally-Extended Prompts Optimization for SAM in Interactive Medical Image Segmentation (paper) | None |
| 202306 | T. Shaharabany et al. | AutoSAM: Adapting SAM to Medical Images by Overloading the Prompt Encoder (paper) | None |
| 202306 | Y. Gao et al. | DeSAM: Decoupling Segment Anything Model for Generalizable Medical Image Segmentation (paper) | Code |
| 202305 | D. Lee et al. | IAMSAM : Image-based Analysis of Molecular signatures using the Segment-Anything Model (paper) | Code |
| 202305 | M. Hu et al. | BreastSAM: A Study of Segment Anything Model for Breast Tumor Detection in Ultrasound Images (paper) | None |
| 202305 | J. Wu | PromptUNet: Toward Interactive Medical Image Segmentation (paper) | Code |
| 202305 | Y. Li et al. | Polyp-SAM: Transfer SAM for Polyp Segmentation (paper) | Code |
| 202305 | C. Mattjie et al. | Exploring the Zero-Shot Capabilities of the Segment Anything Model (SAM) in 2D Medical Imaging: A Comprehensive Evaluation and Practical Guideline (paper) | None |
| 202305 | D. Cheng et al. | SAM on Medical Images: A Comprehensive Study on Three Prompt Modes (paper) | None |
| 202304 | A. Wang et al. | SAM Meets Robotic Surgery: An Empirical Study in Robustness Perspective (paper) | None |
| 202304 | Y. Huang et al. | Segment Anything Model for Medical Images? (paper) | None |
| 202304 | M. Hu et al. | SkinSAM: Empowering Skin Cancer Segmentation with Segment Anything Model (paper) | None |
| 202304 | B. Wang et al. | GazeSAM: What You See is What You Segment (paper) | Code |
| 202304 | K. Zhang and D. Liu | Customized Segment Anything Model for Medical Image Segmentation (paper) | Code |
| 202304 | Z. Qiu et al. | Learnable Ophthalmology SAM (paper) | Code |
| 202304 | P. Shi et al. | Generalist Vision Foundation Models for Medical Imaging: A Case Study of Segment Anything Model on Zero-Shot Medical Segmentation (paper) | None |
| 202304 | J. Wu et al. | Medical SAM Adapter: Adapting Segment Anything Model for Medical Image Segmentation (paper) | Code |
| 202304 | J. Ma and B. Wang | Segment Anything in Medical Images (paper) | Code |
| 202304 | Y. Zhang et al. | Input Augmentation with SAM: Boosting Medical Image Segmentation with Segmentation Foundation Model (paper) | None |
| 202304 | MA. Mazurowski et al. | Segment Anything Model for Medical Image Analysis: an Experimental Study (paper) | Code |
| 202304 | S. He et al. | Accuracy of Segment-Anything Model (SAM) in medical image segmentation tasks (paper) | None |
| 202304 | T. Chen et al. | SAM Fails to Segment Anything? – SAM-Adapter: Adapting SAM in Underperformed Scenes: Camouflage, Shadow, Medical Image Segmentation, and More (paper) | Code |
| 202304 | C. Hu and X. Li | When SAM Meets Medical Images: An Investigation of Segment Anything Model (SAM) on Multi-phase Liver Tumor Segmentation (paper) | None |
| 202304 | F. Putz et al. | The “Segment Anything” foundation model achieves favorable brain tumor autosegmentation accuracy on MRI to support radiotherapy treatment planning (paper) | None |
| 202304 | T. Zhou et al. | Can SAM Segment Polyps? (paper) | Code |
| 202304 | Y. Liu et al. | SAMM (Segment Any Medical Model): A 3D Slicer Integration to SAM (paper) | Code |
| 202304 | S. Roy et al. | SAM.MD: Zero-shot medical image segmentation capabilities of the Segment Anything Model (paper) | None |
| 202304 | S. Mohapatra et al. | SAM vs BET: A Comparative Study for Brain Extraction and Segmentation of Magnetic Resonance Images using Deep Learning (paper) | None |
| 202304 | R. Deng et al. | Segment Anything Model (SAM) for Digital Pathology: Assess Zero-shot Segmentation on Whole Slide Imaging (paper) | None |
7.AutoSAM: Adapting SAM to Medical Images by Overloading the Prompt Encoder
Shaharabany T, Dahan A, Giryes R, et al. AutoSAM: Adapting SAM to Medical Images by Overloading the Prompt Encoder[J]. arXiv preprint arXiv:2306.06370, 2023.
【文章概述】
-
文章介绍了一种名为AutoSAM的方法,旨在改善Segment Anything Model(SAM)在医学影像领域的应用。SAM虽然在图像分割方面有出色的表现,但在处理非自然图像(如医学影像)时效果不佳。AutoSAM的核心思想是替换SAM的条件编码器(prompt encoder)为一个新的prompts生成编码器,这个编码器直接处理输入图像,而不需要进一步微调SAM模型。

图2展示了AutoSAM的框架。在这个框架中,Segment Anything Model (SAM) 的提示编码器(prompt encoder)被替换成了自定义的编码器,而图像编码器(image encoder)和掩码解码器(mask decoder)则保持不变。这个图解说明了如何通过替换SAM的一个组成部分来适应医学图像的处理,而不需要改变其他核心部件。
【方法】
AutoSAM采用了一个辅助的提示编码网络(auxiliary prompt encoder network),通过输入图像生成SAM的替代提示(surrogate prompt)。
- 提示生成网络:
- 网络以图像作为输入,并生成用于SAM的提示,即prompts不是人为输入的,而是选练一个编码器自动生成,AutoSAM其实就是在训练这个prompts生成器,而SAM的图像编码器和mask decoder都不变,从而实现“Auto”
- 这些生成的提示旨在改善SAM在图像分割任务中的表现。
- 训练方法:
- 在训练过程中,SAM网络 会将梯度传播到提示生成网络。
- 梯度传播是基于两种分割损失:二元交叉熵损失(BCE)和Dice损失。
- BCE损失是基于真实mask 和SAM输出 mask的负对数似然,而Dice损失则测量预测mask和真实mask之间的重叠。
【实验】
- 该方法在多个公开的医学图像和视频数据集上进行了评估,表现出比基线方法和其他最先进方法更好的分割性能。
- AutoSAM的训练涉及使用二元交叉熵损失和Dice损失,采用的网络基于Harmonic Dense Net。
- 研究还包括对不同的医学影像数据集进行评估,如MoNuSeg, GlaS, 以及多个息肉(Polyp)数据集。
结果
- AutoSAM在多个医学图像分割基准上取得了最先进的结果,尤其在MoNu和GlaS数据集上的表现显著。
- 在SUN-SEG视频数据集上,AutoSAM即使是作为一个基于图像的方法,也在几乎所有的基准测试中超过了基于视频的方法。
【结论】
- AutoSAM证明了通过为SAM模型提供合适的条件信号,可以有效地改善其在医学图像分割上的性能。
- 未来工作可能包括学习一个通用的AutoSAM网络,用于多个医学成像领域,并探索其在新任务上的泛化能力。
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:veading@qq.com进行投诉反馈,一经查实,立即删除!