数字病理图像分析的开源软件qupath学习 ①
介绍:QuPath是一种新的生物图像分析软件,旨在满足对用户友好、可扩展、开源解决方案日益增长的需求,用于数字病理学和全玻片图像分析。除了提供全面的肿瘤识别和高通量生物标志物评估工具外,QuPath 还为研究人员提供了强大的批处理和脚本功能,以及一个可扩展的平台,用于开发和共享分析复杂组织图像的新算法。此外,QuPath的灵活设计使其适用于生物医学研究中的各种其他图像分析应用。
使用:它提供了第一个全面的开源桌面软件应用程序,专门用于分析和探索整个玻片成像数据。其核心是一个跨平台、多线程、基于图块的全幻灯片图像查看器,其中包含广泛的注释和可视化工具。最重要的是,QuPath提供了一系列新颖的算法,不仅为病理学中常见的、具有挑战性的分析问题提供现成的、用户友好的解决方案,而且还提供了创建自定义工作流程的构建块,并将它们链接在一起,通过强大的脚本功能进行批处理。
文献示例过程:
TMA:从每个病例的肿瘤中心取三个代表性区域进行注释,单个代表性肿瘤块中切割一个新的切片进行H&E染色,注释为组织微阵列(?tissue microarray TMA)。用从供体块中手动穿刺提取直径为1毫米的组织核心(应该是作为TMA)。
免疫组化:CD3 、CD8、p53 和 PD-L1。
扫片:所有 TMA 载玻片均使用 Aperio ScanScope CS 全玻片扫描仪以 40 倍放大倍率扫描,分辨率为 0.25 μm/像素。H&E玻片扫描:231个在Aperio ScanScope扫描仪上扫描,81个在滨松纳米变焦器上扫描,分辨率都在0.231-0.253 μm/像素范围内。
QuPath分析数据:通过使用颜色反卷积分离染色剂并识别平滑后苏木精通道 (CD3) 或苏木精和 DAB 通道总和 (CD8) 中的峰来鉴定单个细胞,并根据平滑的 DAB 通道信息将这些细胞分配为阳性或阴性细胞。使用检测到的阳性细胞数和面积来计算每平方毫米的平均阳性细胞数。设置强度阈值以根据平均核 DAB 光密度进一步将肿瘤细胞细分为 p53 染色的阴性、弱、中度或强阳性。通过添加 3x% 强染色的肿瘤细胞核、2x% 中度染色的肿瘤细胞核和 1x% 弱染色的肿瘤细胞核来计算每个组织核心的 H 评分,给出的结果范围为 0(所有肿瘤核阴性)到 300(所有肿瘤核强阳性)。基质识别:H&E全切片肿瘤间质百分比分析。先用 QuPath 的手动注释工具在所有 312 张 H&E 染色载玻片上注释了具有代表性的肿瘤区域。然后批量应用脚本,自动识别和设置每个图像的红色、绿色和蓝色通道的平均背景强度。然后病理学家用40张切片交互式训练随机树分类器来区分肿瘤上皮、基质和“其他”(空白、粘液、正常肌肉或坏死等)。然后计算肿瘤基质百分比 (TSP) :TSP = AS/(AE + AS) × 100 %
AS 表示归类为基质的总面积,AE 表示归类为上皮的总面积。
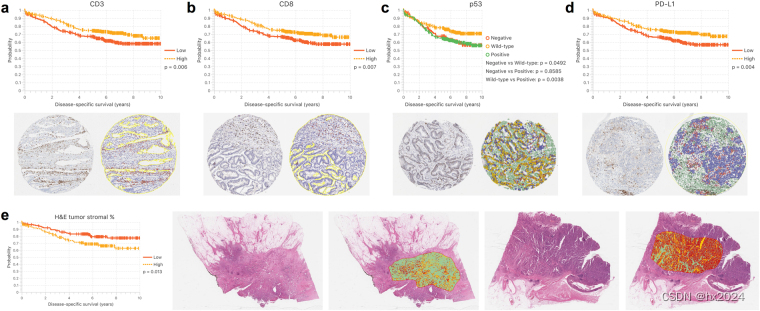
解释:(a-d)对 CD3、CD8、p53 和 PD-L1 染色的 TMA 生物标志物评分进行 Kaplan Meier 生存分析。(e) Kaplan Meier 曲线显示基于中位肿瘤基质百分比的患者分层。代表性图像分别显示了高基质百分比和低基质百分比的肿瘤的原始图像和标记。绿色表示被归类为基质的区域,深红色表示肿瘤上皮,而黄色表示其他分类的组织或空白。
作为深度学习神经网络的训练工具
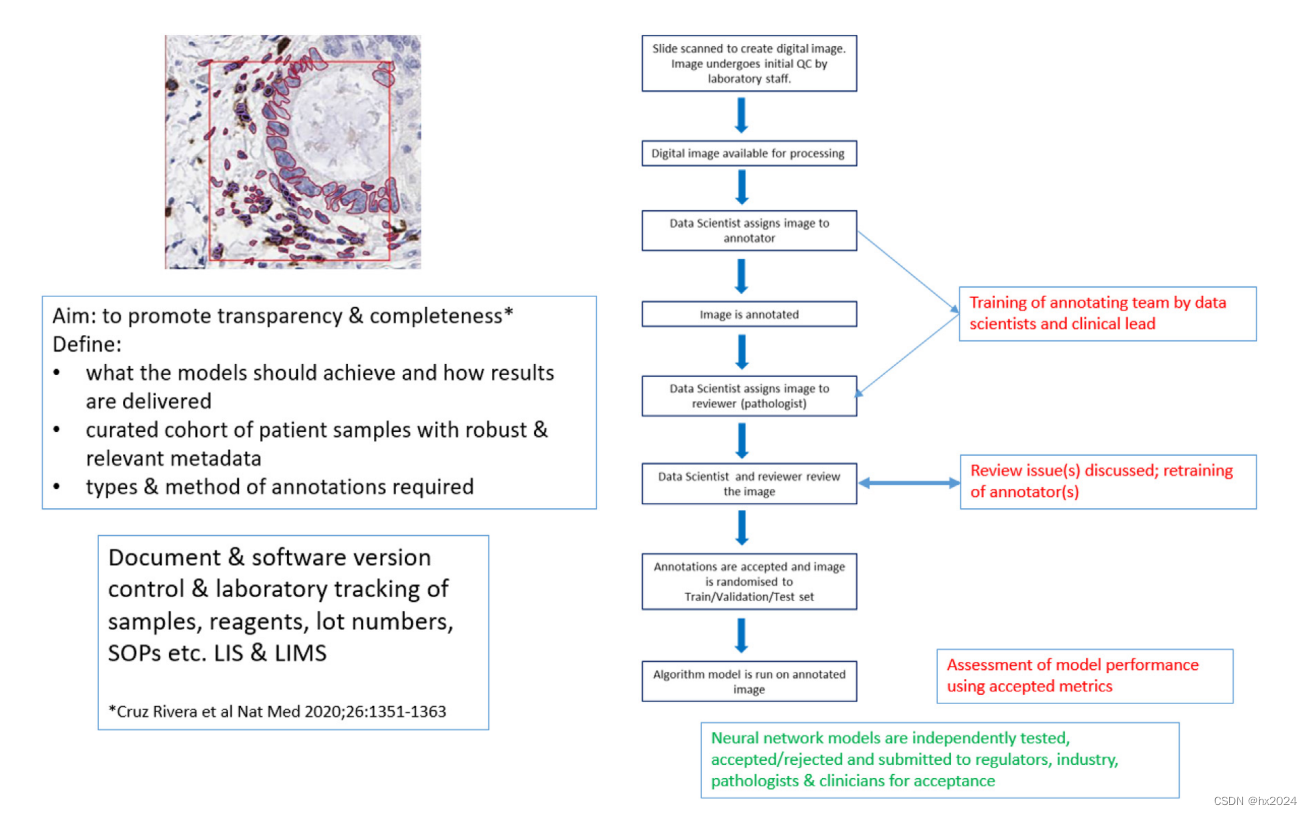
QuPath为高级人工智能的训练、提供和应用提供了一个框架,超越了内置的机器学习方法。该框架可以包括任何内容,从通过病理学家注释或补丁提取来训练更高级的深度学习神经网络,到最终在QuPath之外获取的数据上训练的深度学习模型的可视化。
参考:
1:QuPath: Open source software for digital pathology image analysis - PMC (nih.gov)
2:QuPath: The global impact of an open source digital pathology system
3:qupath/qupath: QuPath - Bioimage analysis & digital pathology (github.com)
4:Projects — QuPath 0.5.0 documentation
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:veading@qq.com进行投诉反馈,一经查实,立即删除!