差分进化算法DE
2023-12-13 21:00:59
此算法是一种基于贪心的并行直接搜索算法。
1.过程
(1)初始化种群
NP个D维的参数向量(i=1,2,...,NP)作为每一代G种群,种群规模必须>=4
(2)变异
使用种群中两个不同向量来干扰一个现有向量,进行差分操作,实现变异。变异时需要确保变异向量不会超出边界,若超出则需要进行修正。F为变异因子∈[0,2],控制差分变化的放大倍数。
对于每个目标向量都要根据三个不同向量生成一个变异向量
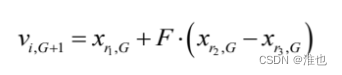

(3)交叉
每个个体和它生成的子代变异向量进行交叉,每个分量按一定的概率选择子代变异向量来生成试验个体,若没有选择子代变异向量则此分量为原个体的分量。按照上述方法生成试验向量。CR为交叉概率因子;jrand是[1,...,D]中随机选择的索引,确保u从v获得至少一个分量。
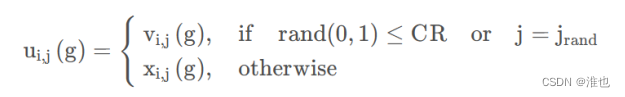

(4)选择
根据贪婪原则,如果试验向量成本函数值<目标向量成本函数值,则下一代的种群个体设置为试验向量,否则保留上一代的目标向量。

2、流程图

3、代码
from random import random
from random import sample
from random import uniform
import matplotlib.pyplot as plt
import matplotlib
matplotlib.use('TkAgg')
# 向量维度的平方求和
def sphere(x):
total = 0
for i in range(len(x)):
total += x[i]**2
return total
# 确保变异向量不超出边界
def ensure_bounds(vec, bounds):
vec_new = []
# 遍历变异向量的每个维度
for i in range(len(vec)):
# 变量比边界最小值还小,赋值为边界最小值
if vec[i] < bounds[i][0]:
vec_new.append(bounds[i][0])
# 变量比边界最大值还大,赋值为边界最大值
if vec[i] > bounds[i][1]:
vec_new.append(bounds[i][1])
# 变量满足边界条件,不进行操作
if bounds[i][0] <= vec[i] <= bounds[i][1]:
vec_new.append(vec[i])
return vec_new
# ——————————————————————————DE————————————————————————————————
def minimize(bounds, popsize, mutate, recombination, maxiter):
# 种群初始化
population = [] # 空的种群
gen_avg_record = [] # 适应度平均值
gen_best_record = [] #适应度最优值
for i in range(0, popsize):
indv = []
for j in range(len(bounds)): # D=2,所以j取0,1
indv.append(uniform(bounds[j][0], bounds[j][1])) # uniform 均匀分布
population.append(indv) # append 追加
# 进行迭代
for i in range(1, maxiter+1):
print("generation:", i) #输出种群
gen_scores = []
for j in range(0, popsize):
# 变异
candidates = list(range(0, popsize))
candidates.remove(j) # 去除目标向量的序号
random_index = sample(candidates, 3) # 从candidates中随机抽取除3个元素
x_1 = population[random_index[0]] # 抽到2,则选取种群中第二个个体作为x_r1g
x_2 = population[random_index[1]]
x_3 = population[random_index[2]]
x_t = population[j] # 目标向量
# x_r2g减x_r3g,生成新向量 x_diff
# zip(numbers, letters) 生成 (x, y) 形式的元组的迭代器,x取自数字.y取自字母
x_diff = [x_2_i - x_3_i for x_2_i,x_3_i in zip(x_2, x_3)]
# 变异向量 v_ig+1 = x_r1g +F * (x_r2g - x_r3g)
v_donor = [x_1_i + mutate * x_diff_i for x_1_i, x_diff_i in zip(x_1, x_diff)]
# 确保变异向量在边界内
v_donor = ensure_bounds(v_donor, bounds)
# 交叉
v_trial = [] # 试验向量
for k in range(len(x_t)):
crossover = random() # 生成[0,1]内的随机数
if crossover <= recombination:
v_trial.append(v_donor[k])
else:
v_trial.append(x_t[k])
# 选择
score_trial = sphere(v_trial) # sphere:向量各个维度的平方和
score_target = sphere(x_t)
if score_trial < score_target:
population[j] = v_trial
gen_scores.append(score_trial)
print(">", score_trial, v_trial)
else:
print(">", score_target, x_t)
gen_scores.append(score_target)
gen_avg = sum(gen_scores) / popsize
gen_best = min(gen_scores)
gen_avg_record.append(gen_avg)
gen_best_record.append(gen_best)
gen_sol = population[gen_scores.index(min(gen_scores))]
print(",种群平均值:", gen_avg)
print(",种群最优值:", gen_best)
print(",最好结果:", gen_sol)
pass
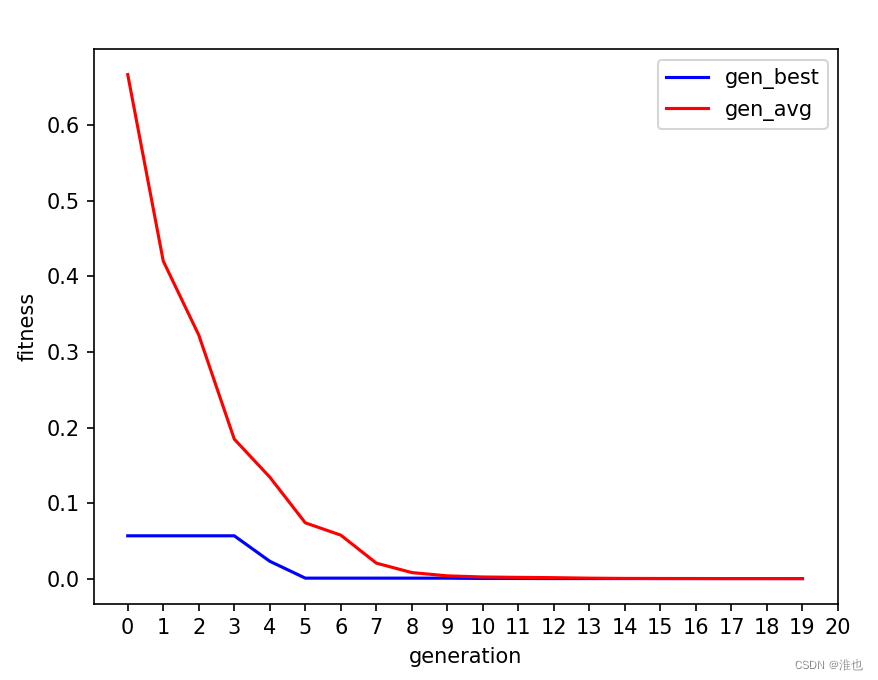
# 画图
plt.plot([i for i in range(len(gen_best_record))], gen_best_record, 'b')
plt.plot([i for i in range(len(gen_avg_record))], gen_avg_record, 'r')
plt.ylabel('fitness')
plt.xlabel('generation')
plt.yticks([0, 0.1, 0.2, 0.3, 0.4, 0.5, 0.6]) # 设置y轴刻度
plt.xticks([0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20]) # 设置x轴刻度
plt.legend(['gen_best', 'gen_avg'])
plt.show()
#设置参数
bounds = [(-1,1),(-1,1)] # 维度D=2,搜索空间范围也叫约束边界
popsize = 10 # 种群规模NP
mutate = 0.5 # 变异因子 F
recombination = 0.7 # 交叉概率 CR [0,1]
maxiter = 20 # 最大迭代次数
# DE/rand/1/bin
minimize(bounds, popsize, mutate, recombination, maxiter)
文章来源:https://blog.csdn.net/qq_51165184/article/details/134975267
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:veading@qq.com进行投诉反馈,一经查实,立即删除!
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。 如若内容造成侵权/违法违规/事实不符,请联系我的编程经验分享网邮箱:veading@qq.com进行投诉反馈,一经查实,立即删除!